Sviluppo di software scientifico
Scientific software development.
Parte 2: Aspetti Pratici con Python
In questo articolo seguiremo i principi del TDD per lo sviluppo di software scientifico come descritto nel primo capitolo di questa serie per sviluppare un filtro di rilevamento dei bordi noto come filtro Sobel.
Nel primo articolo abbiamo parlato di quanto sia importante – e complicato – sviluppare metodi di testing affidabili per il software che spesso presenta problemi complessi nei software scientifici. Abbiamo anche visto come superare questi problemi seguendo un ciclo di sviluppo ispirato al TDD, ma adattato al calcolo scientifico. Riproduco qui di seguito una versione abbreviata di queste istruzioni.
Ciclo di implementazione
- Raccogliere i requisiti
- Progettare lo schema
- Implementare i test iniziali
- Implementare la versione alpha
- Costruire una libreria di oracoli
- Rivedere i test
- Implementare il profiling
Ciclo di ottimizzazione
- Ottimizzare
- Rimplementare
Ciclo di nuovi metodi
- Implementare il nuovo metodo
- Convalidare rispetto agli oracoli precedentemente curati
Per Iniziare: Il Filtro Sobel
In questo articolo, seguiremo le istruzioni sopra riportate per sviluppare una funzione che applica il filtro Sobel. Il filtro Sobel è uno strumento di visione artificiale comunemente utilizzato per rilevare o migliorare i bordi delle immagini. Continua a leggere per vedere alcuni esempi!
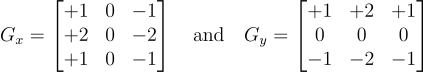
Iniziamo con il primo passo, raccogliamo alcuni requisiti. Seguiremo la formulazione standard del filtro Sobel descritta in questo articolo. In poche parole, l’operatore Sobel consiste nella convoluzione dell’immagine A con i seguenti due kernel 3 × 3, elevando al quadrato ogni pixel delle immagini risultanti, sommandoli e prendendo la radice quadrata punto per punto. Se Ax e Ay sono le immagini risultanti dalle convoluzioni, il risultato del filtro Sobel S è √(Ax² + Ay²).
- Il Calcolo Quantistico Potrebbe Ottenere un Impulso dalla Scoperta del Q-Silicio
- Trasistori di Legno Radicano
- Amministrazione Biden valuta ulteriori restrizioni sulle vendite di chip di intelligenza artificiale alla Cina
Requisiti
Vogliamo che questa funzione prenda un array 2D e generi un altro array 2D. Potremmo volerlo applicare su due assi qualsiasi di un ndarray. Potremmo persino volerlo utilizzare su più (o meno) di due assi. Potremmo avere specifiche su come gestire i bordi dell’array.
Per ora ricordiamoci di mantenerlo semplice e iniziamo con un’implementazione 2D. Ma prima facciamo uno schizzo del design.
Schizzo del Design
Iniziamo con un design semplice, sfruttando le annotazioni di Python. Consiglio vivamente di annotare il più possibile e di utilizzare mypy come linter.
from typing import Tuplefrom numpy.core.multiarray import normalize_axis_indexfrom numpy.typing import NDArraydef sobel(arr: NDArray, axes: Tuple[int, int] = (-2, -1)) -> NDArray: """Calcola il filtro Sobel di un'immagine Parametri ---------- arr : NDArray Immagine di input axes : Tuple[int, int], opzionale Assi su cui calcolare il filtro, per impostazione predefinita (-2, -1) Returns ------- NDArray Output """ # Accetta solo array 2D if arr.ndim != 2: raise NotImplementedError # Assicurati che l'asse[0] sia il primo asse e l'asse[1] sia il secondo # asse. L'oscuro `normalize_axis_index` converte gli indici negativi in # indici compresi tra 0 e arr.ndim - 1. if any( normalize_axis_index(ax, arr.ndim) != i for i, ax in zip(range(2), axes) ): raise NotImplementedError passImplementare i test
Questa funzione non fa molto. Ma è documentata, annotata e le sue limitazioni attuali sono incorporate in essa. Ora che abbiamo un design, passiamo immediatamente ai test.
Prima di tutto, notiamo che le immagini vuote (tutte zeri) non hanno bordi. Quindi devono restituire anche zeri. Infatti, qualsiasi immagine costante dovrebbe restituire anche zeri. Includiamo questi aspetti nei nostri primi test. Vedremo anche come utilizzare il monkey testing per testare molti array.
# test_zero_constant.pyimport numpy as npimport pytest# Test multipli dtypes [email protected]( "dtype", ["float16", "float32", "float64", "float128"],)def test_zero(dtype): # Imposta il seed casuale seed = int(np.random.rand() * (2**32 - 1)) np.random.seed(seed) # Crea un array 2D di forma casuale e riempilo di zeri nx, ny = np.random.randint(3, 100, size=(2,)) arr = np.zeros((nx, ny), dtype=dtype) # Applica la funzione sobel arr_sob = sobel(arr) # `assert_array_equal` dovrebbe fallire nella maggior parte dei casi. # Funzionerà solo quando `arr_sob` è identicamente zero, # il che di solito non è il caso. # NON UTILIZZARE! # np.testing.assert_array_equal( # arr_sob, 0.0, err_msg=f"{seed=} {nx=}, {ny=}" # ) # `assert_almost_equal` può fallire quando usato con decimali elevati. # Si basa anche sul controllo float64, che potrebbe fallire per # tipi float128. # NON UTILIZZARE! # np.testing.assert_almost_equal( # arr_sob, # np.zeros_like(arr), # err_msg=f"{seed=} {nx=}, {ny=}", # decimal=4, # ) # `assert_allclose` con tolleranza personalizzata è il mio metodo preferito # Il 10 è arbitrario e dipende dal problema. Se un metodo # che sai essere corretto non passa, aumenta a 100, ecc. # Se la tolleranza necessaria per superare i test è troppo alta, # assicurati che il metodo sia effettivamente corretto. tol = 10 * np.finfo(arr.dtype).eps err_msg = f"{seed=} {nx=}, {ny=} {tol=}" # Registra i seed e altre informazioni np.testing.assert_allclose( arr_sob, np.zeros_like(arr), err_msg=err_msg, atol=tol, # rtol è inutile per desired=zeros )@pytest.mark.parametrize( "dtype", ["float16", "float32", "float64", "float128"])def test_constant(dtype): seed = int(np.random.rand() * (2**32 - 1)) np.random.seed(seed) nx, ny = np.random.randint(3, 100, size=(2,)) constant = np.random.randn(1).item() arr = np.full((nx, ny), fill_value=constant, dtype=dtype) arr_sob = sobel(arr) tol = 10 * np.finfo(arr.dtype).eps err_msg = f"{seed=} {nx=}, {ny=} {tol=}" np.testing.assert_allclose( arr_sob, np.zeros_like(arr), err_msg=err_msg, atol=tol, # rtol è inutile per desired=zeros )Questo frammento di codice può essere eseguito da linea di comando con
$ pytest -qq -s -x -vv --durations=0 test_zero_constant.pyVersione Alpha
Ovviamente i nostri test attualmente falliranno, ma sono sufficienti per ora. Implementiamo una prima versione.
from typing import Tupleimport numpy as npfrom numpy.core.multiarray import normalize_axis_indexfrom numpy.typing import NDArraydef sobel(arr: NDArray, axes: Tuple[int, int] = (-2, -1)) -> NDArray: if arr.ndim != 2: raise NotImplementedError if any( normalize_axis_index(ax, arr.ndim) != i for i, ax in zip(range(2), axes) ): raise NotImplementedError # Definiamo i nostri kernel di filtro. Nota che ereditano il tipo di input, quindi # un input float32 non deve mai essere convertito in float64 per i calcoli. # Ma riesci a vedere dove l'uso di un altro dtype per Gx e Gy potrebbe avere # senso per alcuni dtype di input? Gx = np.array( [[-1, 0, 1], [-2, 0, 2], [-1, 0, 1]], dtype=arr.dtype, ) Gy = np.array( [[-1, -2, -1], [0, 0, 0], [1, 2, 1]], dtype=arr.dtype, ) # Crea l'array di output e riempilo di zeri s = np.zeros_like(arr) for ix in range(1, arr.shape[0] - 1): for iy in range(1, arr.shape[1] - 1): # Moltiplicazione punto per punto seguita da somma, nota anche come convoluzione s1 = (Gx * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() s2 = (Gy * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() s[ix, iy] = np.hypot(s1, s2) # np.sqrt(s1**2 + s2**2) return sCon questa nuova funzione, tutti i nostri test dovrebbero passare e dovremmo ottenere un output come questo:
$ pytest -qq -s -x -vv --durations=0 test_zero_constant.py........======================================== durate più lunghe =========================================0.09s chiamata t_049988eae7f94139a7067f142bf2852f.py::test_constant[float16]0.08s chiamata t_049988eae7f94139a7067f142bf2852f.py::test_zero[float64]0.06s chiamata t_049988eae7f94139a7067f142bf2852f.py::test_constant[float128]0.04s chiamata t_049988eae7f94139a7067f142bf2852f.py::test_zero[float128]0.04s chiamata t_049988eae7f94139a7067f142bf2852f.py::test_constant[float64]0.02s chiamata t_049988eae7f94139a7067f142bf2852f.py::test_constant[float32]0.01s chiamata t_049988eae7f94139a7067f142bf2852f.py::test_zero[float16](17 durate < 0.005s nascoste. Utilizza -vv per mostrare queste durate.)8 passati in 0.35sFino ad ora abbiamo:
- Raccolto i requisiti del nostro problema.
- Schizzato una progettazione iniziale.
- Implementato alcuni test.
- Implementato la versione alpha, che supera questi test.
Questi test forniscono una verifica per le future versioni, oltre a una libreria di oracoli molto rudimentale. Ma mentre i test unitari sono ottimi per catturare deviazioni minime dai risultati attesi, non sono ottimi nel differenziare risultati errati da risultati quantitativamente diversi – ma comunque corretti. Supponiamo che domani vogliamo implementare un altro operatore di tipo Sobel, che ha un kernel più lungo. Non ci aspettiamo che i risultati corrispondano esattamente alla nostra versione attuale, ma ci aspettiamo che le uscite di entrambe le funzioni siano qualitativamente molto simili.
Inoltre, è una pratica eccellente provare molte diverse voci nelle nostre funzioni per capire come si comportano per diverse voci. Questo assicura che convalidiamo scientificamente i risultati.
Scikit-image ha una eccellente libreria di immagini, che possiamo utilizzare per creare oracoli.
# !pip installscikit-image poochfrom typing import Dict, Callableimport numpy as npimport skimage.databwimages: Dict[str, np.ndarray] = {}for attrname in skimage.data.__all__: attr = getattr(skimage.data, attrname) # I dati vengono ottenuti tramite chiamate di funzione if isinstance(attr, Callable): try: # Scarica i dati data = attr() # Assicurati che sia un array 2D if isinstance(data, np.ndarray) and data.ndim == 2: # Converti da vari tipi di int a float32 per valutare meglio la precisione bwimages[attrname] = data.astype(np.float32) except: continue# Applica sobel alle immaginibwimages_sobel = {k: sobel(v) for k, v in bwimages.items()}Una volta che abbiamo i dati, possiamo tracciarli.
import matplotlib.pyplot as pltfrom mpl_toolkits.axes_grid1 import make_axes_locatabledef create_colorbar(im, ax): divider = make_axes_locatable(ax) cax = divider.append_axes("right", size="5%", pad=0.1) cb = ax.get_figure().colorbar(im, cax=cax, orientation="vertical") return cax, cbfor name, data in bwimages.items(): fig, axs = plt.subplots( 1, 2, figsize=(10, 4), sharex=True, sharey=True ) im = axs[0].imshow(data, aspect="equal", cmap="gray") create_colorbar(im, axs[0]) axs[0].set(title=name) im = axs[1].imshow(bwimages_sobel[name], aspect="equal", cmap="gray") create_colorbar(im, axs[1]) axs[1].set(title=f"{name} sobel")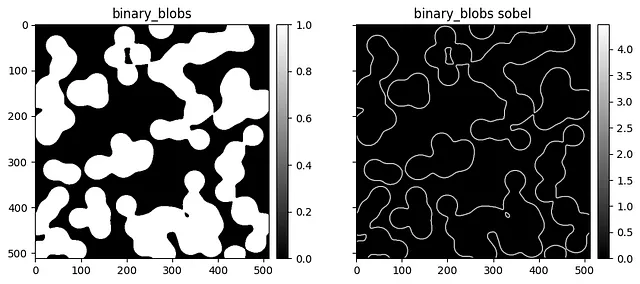

Queste sembrano davvero buone! Consiglio di conservarle, sia come array che come figure, che posso controllare rapidamente per una nuova versione. Consiglio vivamente HD5F per la memorizzazione degli array. Puoi anche configurare una Sphinx Gallery per generare direttamente le figure quando si aggiornano i documenti, in modo da avere una costruzione riproducibile delle figure che puoi utilizzare per confrontare con le versioni precedenti.
Dopo che i risultati sono stati convalidati, puoi memorizzarli su disco e utilizzarli come parte dei tuoi test di unità. Qualcosa del genere:
oracle_library = [(k, v, bwimages_sobel[k]) for k, v in bwimages.items()]# save_to_disk(oracle_library, ...)
# test_oracle.pyimport numpy as npimport pytestfrom numpy.typing import NDArray# oracle_library = read_from_disk(...)@pytest.mark.parametrize("name,input,output", oracle_library)def test_oracles(name: str, input: NDArray, output: NDArray): output_new = sobel(input) tol = 10 * np.finfo(input.dtype).eps mean_avg_error = np.abs(output_new - output).mean() np.testing.assert_allclose( output_new, output, err_msg=f"{name=} {tol=} {mean_avg_error=}", atol=tol, rtol=tol, )Profilazione
Calcolare il filtro di Sobel per questi dataset ha richiesto del tempo! Quindi il prossimo passo è profilare il codice. Consiglio di creare un file benchmark_xyz.py per ogni test e rieseguirli regolarmente per individuare eventuali regressioni delle prestazioni. Questo può anche far parte dei tuoi test di unità, ma non andremo così lontano in questo esempio. Un’altra idea è utilizzare gli oracoli sopra per il benchmark.
Ci sono molti modi per misurare il tempo di esecuzione del codice. Per ottenere il tempo trascorso effettivo a livello di sistema, utilizza perf_counter_ns dal modulo integrato time per misurare il tempo in nanosecondi. In un notebook Jupyter, la magia di cella integrata %%timeit cronometra l’esecuzione di una determinata cella. Il decoratore qui sotto si ispira a questa magia di cella e può essere utilizzato per misurare il tempo di qualsiasi funzione.
import timefrom functools import wrapsfrom typing import Callable, Optionaldef sizeof_fmt(num, suffix="s"): for unit in ["n", "μ", "m"]: if abs(num) < 1000: return f"{num:3.1f} {unit}{suffix}" num /= 1000 return f"{num:.1f}{suffix}"def timeit( func_or_number: Optional[Callable] = None, number: int = 10,) -> Callable: """Applicare a una funzione per misurarne le esecuzioni. Parametri ---------- func_or_number : Optional[Callable], opzionale Funzione da decorare o argomento `number` (vedi sotto), per default None number : int, opzionale Numero di volte che la funzione verrà eseguita per ottenere le statistiche, per default 10 Returns ------- Callable Quando viene fornita una funzione, restituisce la funzione decorata. Altrimenti restituisce un decoratore. Esempi -------- .. code-block:: python @timeit def my_fun(): pass @timeit(100) def my_fun(): pass @timeit(number=3) def my_fun(): pass """ if isinstance(func_or_number, Callable): func = func_or_number number = number elif isinstance(func_or_number, int): func = None number = func_or_number else: func = None number = number def decorator(f): @wraps(f) def wrapper(*args, **kwargs): runs_ns = np.empty((number,)) # Esegui la prima volta e misura il risultato start_time = time.perf_counter_ns() result = f(*args, **kwargs) runs_ns[0] = time.perf_counter_ns() - start_time for i in range(1, number): start_time = time.perf_counter_ns() f(*args, **kwargs) # Senza memorizzazione, più veloce runs_ns[i] = time.perf_counter_ns() - start_time time_msg = f"{sizeof_fmt(runs_ns.mean())} ± " time_msg += f"{sizeof_fmt(runs_ns.std())}" print( f"{time_msg} per esecuzione (media ± deviazione standard di {number} esecuzioni)" ) return result return wrapper if func is not None: return decorator(func) return decoratorMetto alla prova la nostra funzione:
arr_test = np.random.randn(500, 500)sobel_timed = timeit(sobel)sobel_timed(arr_test);# 3.9s ± 848.9 ms per run (media ± dev. std. di 10 esecuzioni)Non molto veloce 🙁
Quando si indaga la lentezza o le regressioni delle prestazioni, è anche possibile tracciare il tempo di esecuzione di ogni riga. La libreria line_profiler è un ottimo strumento per questo. Può essere utilizzata tramite la cella magica di Jupyter, o utilizzando l’API. Ecco un esempio di utilizzo dell’API:
from line_profiler import LineProfilerlp = LineProfiler()lp_wrapper = lp(sobel)lp_wrapper(arr_test)lp.print_stats(output_unit=1) # 1 per secondi, 1e-3 per millisecondi, ecc.Ecco un esempio di output:
Unità di misura del timer: 1 sTempo totale: 4.27197 sFile: /tmp/ipykernel_521529/1313985340.pyFunzione: sobel alla riga 8Linea # Colpi Tempo Per Colpo % Tempo Contenuto della linea============================================================== 8 def sobel(arr: NDArray, axes: Tuple[int, int] = (-2, -1)) -> NDArray: 9 # Accetta solo array 2D 10 1 0.0 0.0 0.0 if arr.ndim != 2: 11 raise NotImplementedError 12 13 # Assicurarsi che l'asse[0] sia il primo asse e l'asse[1] sia il secondo 14 # asse. L'oscura funzione `normalize_axis_index` converte gli indici negativi in 15 # indici compresi tra 0 e arr.ndim - 1. 16 1 0.0 0.0 0.0 if any( 17 normalize_axis_index(ax, arr.ndim) != i 18 1 0.0 0.0 0.0 for i, ax in zip(range(2), axes) 19 ): 20 raise NotImplementedError 21 22 1 0.0 0.0 0.0 Gx = np.array( 23 1 0.0 0.0 0.0 [[-1, 0, 1], [-2, 0, 2], [-1, 0, 1]], 24 1 0.0 0.0 0.0 dtype=arr.dtype, 25 ) 26 1 0.0 0.0 0.0 Gy = np.array( 27 1 0.0 0.0 0.0 [[-1, -2, -1], [0, 0, 0], [1, 2, 1]], 28 1 0.0 0.0 0.0 dtype=arr.dtype, 29 ) 30 1 0.0 0.0 0.0 s = np.zeros_like(arr) 31 498 0.0 0.0 0.0 for ix in range(1, arr.shape[0] - 1): 32 248004 0.1 0.0 2.2 for iy in range(1, arr.shape[1] - 1): 33 248004 1.8 0.0 41.5 s1 = (Gx * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() 34 248004 1.7 0.0 39.5 s2 = (Gy * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() 35 248004 0.7 0.0 16.8 s[ix, iy] = np.hypot(s1, s2) 36 1 0.0 0.0 0.0 return sMolto tempo viene speso all’interno del ciclo più interno. NumPy preferisce la matematica vettorializzata, poiché può fare affidamento sul codice compilato. Dal momento che stiamo utilizzando cicli for espliciti, ha senso che questa funzione sia molto lenta.
Inoltre, è importante essere intelligenti riguardo alle allocazioni di memoria all’interno dei cicli. NumPy è abbastanza intelligente nell’allocazione di piccole quantità di memoria all’interno dei cicli, quindi le righe che definiscono s1 o s2 potrebbero non allocare nuova memoria. Ma potrebbero anche farlo, o potrebbe esserci un’altra allocazione di memoria che avviene sotto il cofano di cui non siamo consapevoli. Raccomando quindi anche il profiling della memoria. Mi piace usare Memray per questo, ma ci sono anche altri come Fil e Sciagraph. Eviterei anche memory_profiler che (purtroppo!) non viene più mantenuto. Inoltre, Memray è molto più potente. Ecco un esempio di Memray in azione:
$ # Creare sobel.bin che contiene le informazioni di profilazione$ memray run -fo sobel.bin --trace-python-allocators sobel_run.pyScrittura dei risultati del profilo in sobel.binMemray ATTENZIONE: Correzione del simbolo per aligned_alloc da 0x7fc5c984d8f0 a 0x7fc5ca4a5ce0[memray] Risultati del profilo generati con successo.Ora è possibile generare rapporti dai record di allocazione memorizzati.Alcuni comandi di esempio per generare rapporti:python3 -m memray flamegraph sobel.bin
$ # Generare un grafico a fiamma$ memray flamegraph -fo sobel_flamegraph.html --temporary-allocations sobel.bin⠙ Calcolo della linea di galleggiamento più alta... ━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╸ 99% 0:00:0100:01⠏ Elaborazione dei record di allocazione... ━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╸ 98% 0:00:0100:01Scritto sobel_flamegraph.html
$ # Mostra l'albero di memoria$ memray tree --temporary-allocations sobel.bin⠧ Calcolo della linea di galleggiamento più alta... ━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╸ 100% 0:00:0100:01⠧ Elaborazione dei record di allocazione... ━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╸ 100% 0:00:0100:01Metadati di allocazione-------------------Argomenti della riga di comando: 'memray run -fo sobel.bin --trace-python-allocators sobel_run.py'Dimensione massima della memoria: 11.719MBNumero di allocazioni: 15332714Le 10 allocazioni più grandi:-----------------------📂 123.755MB (100.00 %) <ROOT> └── [[3 frame nascosti in 2 file]] └── 📂 123.755MB (100.00 %) _run_code /usr/lib/python3.10/runpy.py:86 ├── 📂 122.988MB (99.38 %) <module> sobel_run.py:40 │ ├── 📄 51.087MB (41.28 %) sobel sobel_run.py:35 │ ├── [[1 frame nascosto in 1 file]] │ │ └── 📄 18.922MB (15.29 %) _sum │ │ lib/python3.10/site-packages/numpy/core/_methods.py:49 │ └── [[1 frame nascosto in 1 file]] │ └── 📄 18.921MB (15.29 %) _sum │ lib/python3.10/site-packages/numpy/core/_methods.py:49...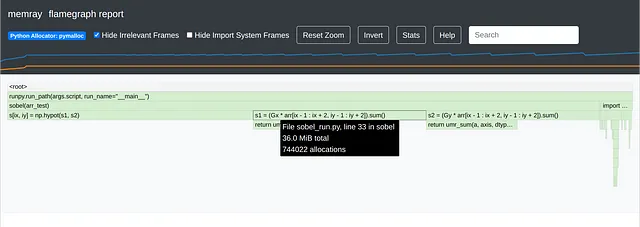
Versione Beta e un Bug
Ora che abbiamo una versione alpha funzionante e alcune funzioni di profilazione, sfrutteremo la libreria SciPy per ottenere una versione molto più veloce.
from typing import Tupleimport numpy as npfrom numpy.core.multiarray import normalize_axis_indexfrom numpy.typing import NDArrayfrom scipy.signal import convolve2ddef sobel_conv2d( arr: NDArray, axes: Tuple[int, int] = (-2, -1)) -> NDArray: if arr.ndim != 2: raise NotImplementedError if any( normalize_axis_index(ax, arr.ndim) != i for i, ax in zip(range(2), axes) ): raise NotImplementedError # Creazione dei kernel come un singolo array complesso. Ci permette di utilizzare # np.abs invece di np.hypot per calcolare la magnitudine. G = np.array( [[-1, 0, 1], [-2, 0, 2], [-1, 0, 1]], dtype=arr.dtype, ) G = G + 1j * np.array( [[-1, -2, -1], [0, 0, 0], [1, 2, 1]], dtype=arr.dtype, ) s = convolve2d(arr, G, mode="same") np.absolute(s, out=s) # In-place abs return s.real
sobel_timed = timeit(sobel_conv2d)sobel_timed(arr_test)# 14.3 ms ± 1.71 ms per loop (mean ± std. dev. of 10 runs)Molto meglio! Ma è corretto?
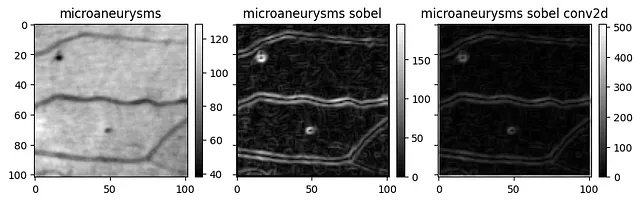
Le immagini sembrano molto simili, ma se si osserva la scala dei colori, non sono uguali. Eseguendo i test viene segnalato un piccolo errore medio. Fortunatamente, ora siamo ben equipaggiati per rilevare differenze quantitative e qualitative.
Dopo aver indagato su questo bug, lo attribuiamo alle diverse condizioni di contorno. Consultando la documentazione di convolve2d, scopriamo che l’array di input viene riempito con zeri prima di applicare il kernel. Nella versione alpha, abbiamo riempito l’output!
Quale delle due è corretta? Argomentativamente, l’implementazione di SciPy ha più senso. In questo caso dovremmo adottare la nuova versione come oracolo e, se necessario, correggere la versione alpha per farla corrispondere. Questo è comune nello sviluppo di software scientifico: nuove informazioni su come fare le cose meglio cambiano gli oracoli e i test.
In questo caso, la correzione è semplice, riempiamo l’array con zeri prima di elaborarlo.
def sobel_v2(arr: NDArray, axes: Tuple[int, int] = (-2, -1)) -> NDArray: # ... arr = np.pad(arr, (1,)) # Dopo il riempimento, ha forma (nx + 2, ny + 2) s = np.zeros_like(arr) for ix in range(1, arr.shape[0] - 1): for iy in range(1, arr.shape[1] - 1): s1 = (Gx * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() s2 = (Gy * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() s[ix - 1, iy - 1] = np.hypot(s1, s2) # Regoliamo gli indici return sUna volta corretta la nostra funzione, possiamo aggiornare gli oracoli e i test che si basano su di essa.
Considerazioni Finali
Abbiamo visto come mettere in pratica alcune delle idee di sviluppo software esplorate in questo articolo. Abbiamo anche visto alcuni strumenti che puoi utilizzare per sviluppare codice di alta qualità e ad alte prestazioni.
Ti suggerisco di provare alcune di queste idee nei tuoi progetti. In particolare, pratica il profiling del codice e miglioralo. La funzione del filtro di Sobel che abbiamo codificato è molto inefficiente, ti suggerisco di cercare di migliorarla.
Ad esempio, prova la parallelizzazione della CPU con un compilatore JIT come Numba, spostando il ciclo interno in Cython o implementando una funzione CUDA GPU con Numba o CuPy. Sentiti libero di dare un’occhiata alla mia serie sulla codifica di kernel CUDA con Numba.






