PyLogik per la de-identificazione dei dati delle immagini mediche.
PyLogik - de-identification tool for medical image data.
Uno strumento di de-identificazione di immagini mediche all’avanguardia e open source

Le raccolte di dati sono ora una delle nostre risorse più preziose. L’informazione come merce non è un concetto nuovo, ma il nostro mondo del 21° secolo è molto diverso da quello di prima. La corsa all’IA è iniziata e in parallelo si sviluppano gli strumenti e le risorse di cui abbiamo bisogno per facilitarla. Raggruppare le informazioni per creare inferenze che siano robuste, generalizzabili e utili per il nostro quotidiano è molto più difficile di quanto sembri. Ciò è particolarmente vero per i dati medici. I dati medici soffrono di una miriade di difficoltà quando si tratta di estrarre valore in modo aggregato e nello sviluppo di algoritmi. Oltre ad essere rumorosi, sono strettamente regolamentati dalle istituzioni e dai centri di cura che agiscono come custodi dei dati (e per una buona ragione), poiché contengono informazioni personali sulla salute (PHI).
PHI, se divulgati, potrebbero avere effetti dannosi sulla privacy delle persone. Questi potrebbero variare da una semplice imbarazzante discriminazione sul posto di lavoro/o con compagnie assicurative fino al furto di identità. Pertanto, quando i ricercatori e le istituzioni accettano di raggruppare le informazioni per creare raccolte, devono esistere un accordo sull’utilizzo dei dati e strumenti per de-identificare i dati il più possibile. PHI può assumere diverse forme: Dirette: numeri di assistenza sanitaria, SSN e date di nascita, anche se i nomi non sono tecnicamente unici, sono trattati come tali. Ci sono anche quasi identificatori, come la data in cui l’immagine è stata raccolta. Inoltre, quando si addestra un algoritmo di apprendimento automatico, questi nomi potrebbero essere considerati informazioni inutili che non vogliamo, infatti, apprendere. Pertanto, rimuoverli è necessario per molteplici motivi.
Ci sono stati numerosi tentativi di effettuare la de-identificazione delle immagini mediche. Sfortunatamente, le soluzioni proposte hanno sofferto di una mancanza di successo, sono specifiche per il sistema operativo o sono disponibili solo a pagamento attraverso fornitori o ricercatori proprietari [1-4]. L’altro problema che ho notato leggendo questi studi è che i ricercatori hanno cercato di mascherare/rimuovere SOLO il testo identificativo e mantenere il testo utile ma scientifico integro. E sebbene questo sia un obiettivo nobile, questo è un problema DAVVERO difficile. Ciò è dovuto al fatto che PHI può assumere formati diversi a seconda del fornitore di attrezzature e del sistema ospedaliero. Vogliamo mantenere le informazioni scientifiche, ma nel caso di una pila di immagini, queste informazioni si ripetono in ogni fotogramma. Ciò significa che consuma spazio ridondante nell’immagine, quindi se possiamo rimuoverlo una volta, possiamo risparmiare spazio di archiviazione! Pertanto, possiamo pensare a queste come ai nostri vincoli di progettazione. Quindi, con questi in mente, risolviamo il problema con tutti questi in mente contemporaneamente.
Employiamo l’apprendimento automatico sotto forma di una rete neurale convoluzionale ricorrente. Per iniziare la pipeline, il primo compito è identificare, estrarre e mascherare i dati basati su testo trovati nelle matrici. Questo processo utilizza PyTorch come framework per la rilevazione del testo. Il modello di riconoscimento del testo si basa su una rete neurale convoluzionale ricorrente (CRNN), che è stata addestrata sui dataset IC13, IIIT5k e SVT. Il modello comprende tre componenti chiave:
- Mappatura in avanti e all’indietro per la visione artificiale
- Novo Nordisk supporterà i postdottorati del MIT che lavorano all’intersezione tra intelligenza artificiale e scienze della vita.
- Incontra LieGAN un framework di AI che utilizza l’addestramento generativo avversario per scoprire automaticamente le equivalenze da un dataset.
a) Estrazione di caratteristiche, ottenuta attraverso una combinazione di ResNet (una rete neurale convoluzionale) e la visual geometry group (VGG) neural network. Questo è responsabile del rilevamento di caratteristiche che assomigliano a lettere.
b) Etichettatura della sequenza, realizzata utilizzando una rete a memoria a lungo e breve termine (LSTM). La parte ricorrente di questo è importante per garantire che le caratteristiche che si trovano una accanto all’altra nell’immagine che appaiono come testo siano considerate raggruppate insieme – il che significa che formano una parola (o più parole) coerente.
c) In cima a questo, c’è un classificatore temporale connessionista (CTC) che agisce per eseguire il riconoscimento ottico dei caratteri (OCR). Questo è responsabile della trascrizione della/e parola/e rilevata/e in lettere basandosi su un lessico inglese. Pertanto, la decodifica viene eseguita dal CTC.
Una panoramica sulla pulizia dei dati e l’anonimizzazione implica il caricamento dei dati, che possono essere in forma di DICOM, stack JPEG o file video, e sottoporli a rilevamento del testo e rimozione attraverso una procedura OCR e di mascheramento. Qualsiasi testo identificato viene estratto e salvato in un file .csv separato (sotto). Se l’utente decide di oscurare i nomi dei file, viene concatenata una serie casuale di caratteri alfanumerici e il nome originale del file viene ulteriormente aggiunto al file .csv, che ora serve simultaneamente come file di incrocio. Inoltre, le immagini vengono migliorate eliminando eventuali elementi estranei. La regione di interesse (ROI) viene quindi isolata utilizzando una sequenza di operazioni di filtraggio, morfologiche e geometriche (di cui esistono varie versioni spiegate di seguito).
In questo lavoro, mostriamo la utilità dei nostri algoritmi disponibili e forniamo indicazioni su come gli utenti finali possono integrarli nelle loro rispettive applicazioni. Il nostro software, PyLogik, può essere installato tramite il terminale utilizzando il comando pip install PyLogik. Abbiamo progettato diverse funzioni che possono essere utilizzate insieme o separatamente nella pipeline. Il nostro software supporta vari tipi di immagini, tra cui 2D (scala di grigi), 3D (scala di grigi con più frame o RGB a 3 canali) e 4D (più frame con informazioni RGB) e può leggere tipi di immagine dicom, .png, .jpg, .jpeg e .nii (NIfTi). Qualsiasi file saltato e i relativi dettagli di elaborazione vengono registrati nei file di log nella cartella di destinazione.
Il flusso di lavoro generale del mio programma è il seguente:

Le nostre funzioni possono essere importate e utilizzate nel seguente modo. Installa la libreria utilizzando il terminale:
$ pip install pylogikImporta le librerie:
from pylogik import deidfrom pylogik import im_analysis Ci sono varie funzioni disponibili per l’utente:

Questa metodologia è stata inizialmente progettata intorno alla nozione di de-id’ing delle immagini ad ultrasuoni, ma è stata poi ampliata per includere altre modalità di imaging che potrebbero non voler essere così restrittive sulla parte di “pulizia” dell’immagine. Toccheremo brevemente le varie opzioni.
- Questo è solo per de-id. Rimuove solo il testo bruciato dall’immagine, lo scrive in un file .csv, con il nome del file (per scopi di crosswalk), e scrive i frame dell’immagine in JPEG senza perdita di dati.
deid.deid(input_path = "path_to_files", output_path="path_to_save_files",rename_files = False, threshold = 0)- input_path : percorso dei file immagine (DICOM, JPEG o video)
- output_path : percorso per salvare i nuovi file immagine e i file di testo .csv
- rename_files : False (default) cambia il nome del file in una serie di 10 caratteri alfanumerici casuali
- threshold : 0 (default) valore intero della soglia nell’immagine (default = 0). Se non è chiaro all’utente, può usare il valore predefinito o utilizzare lo strumento di selezione del colore per catturare l’intensità dello sfondo dall’immagine campione
2. Questo è per de-id e pulizia specifica dei dati ad ultrasuoni. Questo è stato in realtà il primo che ho creato, e ha alcune interessanti comparazioni geometriche che vengono eseguite solo per mantenere una ROI molto pulita. Rimuove il testo bruciato dall’immagine, lo scrive in un file .csv (con il nome del file per scopi di crosswalk), elabora e comprime le immagini secondo i metodi descritti nel paper associato e scrive i frame dell’immagine in JPEG senza perdita di dati.
deid.deid_us(input path = "path_to_files", output path="path_to_file_save",rename_files=False, thresh=0)- input_path : percorso dei file immagine (DICOM, JPEG o video)
- output_path : percorso per salvare i nuovi file immagine e i file di testo .csv
- rename_files : False (default) cambia il nome del file in una serie di 10 caratteri alfanumerici casuali
- threshold : 0 (default) valore intero della soglia nell’immagine (default = 0). Se non è chiaro all’utente, può usare il valore predefinito o utilizzare lo strumento di selezione del colore per catturare l’intensità dello sfondo dall’immagine campione
3. Questo è per de-id e pulizia, in cui viene salvato solo l’oggetto più saliente dell’immagine. Rimuove il testo bruciato dall’immagine, lo scrive in un file .csv (con il nome del file per scopi di crosswalk), mantiene solo l’oggetto più saliente nell’immagine – comprime di conseguenza e scrive i frame dell’immagine in JPEG senza perdita di dati.
deid.deid_one(input_path = "percorso_ai_file", output_path="percorso_per_salvare_il_file",rename_files=False, threshold = 0)- input_path : percorso ai file immagine (DICOM, JPEG o video)
- output_path : percorso per salvare i nuovi file immagine e i file di testo .csv
- threshold : 0 (predefinito) valore intero della soglia nell’immagine (predefinito = 0). Se non è chiaro all’utente, può utilizzare il valore predefinito o utilizzare lo strumento di selezione del colore per acquisire l’intensità dello sfondo dall’immagine di esempio
- rename_files : False (predefinito) cambia il nome del file in una serie di 10 alfanumerici selezionati casualmente
4. Questo è per la de-identificazione e la pulizia, dove solo gli oggetti piccoli vengono filtrati e molte entità grandi rimarranno nell’immagine. Questo rimuove il testo bruciato dall’immagine, lo scrive in un file .csv, con il nome del file (per scopi di incrocio) e scrive il frame / i frame dell’immagine in JPEG senza perdite (rimuove / estrae il testo e rimuove le funzioni a piccola scala)
deid.deid_clean((input_path = "percorso_ai_file", output_path="percorso_per_salvare_il_file",rename_files=False, threshold = 0)- input_path : percorso ai file immagine (DICOM, JPEG o video)
- output_path : percorso per salvare i nuovi file immagine e i file di testo .csv
- rename_files : False (predefinito) cambia il nome del file in una serie di 10 alfanumerici selezionati casualmente
- threshold : 0 (predefinito) valore intero della soglia nell’immagine (predefinito = 0). Se non è chiaro all’utente, può utilizzare il valore predefinito o utilizzare lo strumento di selezione del colore per acquisire l’intensità dello sfondo dall’immagine di esempio
5. Questo è se si hanno immagini di cui si vuole semplicemente rilevare/leggere il testo in un CSV. Tuttavia, non si desidera produrre alcuna immagine in output. Questa funzione individua solo il testo nell’immagine e lo scrive in una serie di file CSV nella cartella specificata in output, senza produrre immagini.
deid.find_txt(input_path = "percorso_ai_file", output_path="percorso_per_salvare_il_file")- input_path : percorso ai file immagine (DICOM, JPEG o video)
- output_path : percorso per salvare i nuovi file immagine e i file di testo .csv
- thresh: valore intero della soglia nell’immagine (predefinito = 0). Se non è chiaro all’utente, può utilizzare il valore predefinito o utilizzare lo strumento di selezione del colore per acquisire l’intensità dello sfondo dall’immagine di esempio
6. Queste sono alcune funzioni aggiuntive contenute nel pacchetto che potrebbero essere utili per il calcolo e la presentazione dei punteggi di Dice.
A) Calcolo del punteggio di Dice:
im_analysis.dice_score(pred_array, true_array, k=1)- pred — array della segmentazione prevista
- true — array della segmentazione della verità fondamentale
- k — valore per la corrispondenza (predefinito = 1)
- Restituisce: punteggio di Dice (float)

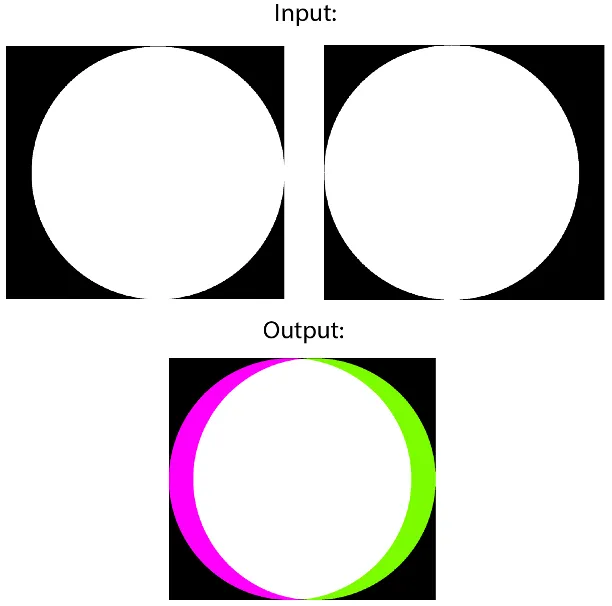
B) Visualizzazione del calcolo di Dice
im_analysis.imshowpair(pred_array, true_array, color1 = (124,252,0), color2 =(255,0,252), show_fig=True)- pred_array — array della segmentazione prevista
- true _array — array della segmentazione della verità fondamentale
- color1 — primo colore per mostrare i valori univoci dalla prima immagine
- color2 — secondo colore per mostrare i valori univoci dalla seconda immagine
- Restituisce: array e grafico
![Immagine sorgente da [1] (questo utilizza la funzione 'deid_us')](https://miro.medium.com/v2/resize:fit:640/format:webp/1*g8nMf2AArjRMSZ5BDwno6w.png)
Al ripensamento degli obiettivi stabiliti nell’introduzione, abbiamo sviluppato con successo un protocollo robusto in termini di deidentificazione, isolamento delle informazioni pertinenti del paziente, identificazione dell’ROI e compressione dei file delle immagini mediche. Il lavoro precedente si è concentrato sulla formazione delle CNN per rilevare e rimuovere solo le informazioni relative all’IPF contenute nell’immagine, con livelli di successo variabili tra il 65% e l’89% [1-3]. Tuttavia, alcune di queste tecniche sono specifiche del sistema operativo o disponibili solo a pagamento [4]. Il pacchetto PyLogik affronta questi problemi garantendo la rimozione degli identificatori diretti dei pazienti mentre converte il formato dei file di testo in un output di file .csv, identificando correttamente l’ROI e compressando le informazioni. Inoltre, il protocollo è OS-agnostic e gratuito per i ricercatori. Semplificando il problema di deep learning e rimuovendo tutto il testo, PyLogik supera il rischio di distinguere caratteri come “Bg” da “B9”, “B1” da “Bl” o “B0” da “Bo”. Ciò consente ai singoli siti di inserire la necessaria filtrazione specifica del contesto sui loro file .csv e garantisce una maggiore efficacia nella rimozione dell’IPF. PyLogik può essere eseguito su qualsiasi sistema operativo ed è gratuito da scaricare, consentendo così di eseguirlo su server dietro i firewall istituzionali. Estraendo e successivamente mascherando tutto il testo, i file .csv prodotti dal pipeline consentono agli utenti finali di interrogare, includere o distruggere informazioni per i loro usi specifici. La nostra strategia facilita anche una migliore integrazione multimodale delle informazioni sui dati. Ad esempio, nelle immagini ecocardiografiche, la frequenza cardiaca viene spesso visualizzata come testo in ogni vista; in PyLogik, queste informazioni vengono conservate e messe a disposizione degli utenti finali, rendendole disponibili per l’uso durante la fusione delle informazioni (precoce, congiunta e tardiva) nello sviluppo dell’algoritmo [5]. Le immagini vengono salvate e prodotte come stack JPEG per ridurre il numero di librerie specializzate e piattaforme di codice necessarie per importare nuovamente le immagini per l’elaborazione [6]. Troncando l’immagine per contenere solo l’ROI (i), conserviamo solo le informazioni salienti, facilitando così la compressione su server secondari non PACS.

Oltre a fornire un protocollo efficiente di deidentificazione e pulizia delle immagini per facilitare l’utilizzo delle immagini a ultrasuoni in modo aggregato per lo sviluppo di algoritmi, il nostro metodo proposto offre una compressione fino al 72% rispetto ai file DICOM originali. Ciò non solo ha implicazioni per la conservazione a lungo termine di questi grandi file, ma consente anche di aumentare notevolmente la conservazione a breve termine per le applicazioni di apprendimento automatico (ovvero, elaborazione batch). Queste immagini vengono salvate come JPEGS senza perdita di dati, dove “senza perdita di dati” significa che l’ROI salvata ha la stessa risoluzione spaziale presente nel formato immagine originale. Questo pacchetto è progettato per essere modulare, con una classe separata per coloro che cercano solo la procedura di deidentificazione della pipeline. Questa parte dell’elaborazione della pipeline può essere facilmente estesa ad altre modalità di imaging come la risonanza magnetica (MRI), la tomografia computerizzata (CT) e altre radiografie, ecc. Il nostro programma fornisce un algoritmo di deidentificazione all’avanguardia (SOTA) applicabile a molteplici modalità di imaging medico, offrendo contemporaneamente la compressione delle immagini (fino al 72% in meno) e preparando i dati per gli esperimenti di apprendimento automatico. Questa compressione è importante poiché ha implicazioni per la conservazione basata su cloud a lungo termine e per la memoria durante l’addestramento degli algoritmi di apprendimento automatico e non viene discussa in altre pubblicazioni di questo tipo. A questo scopo, abbiamo sviluppato una libreria Python open-source, PyLogik. È facile da installare, agnostico del sistema operativo, può essere eseguito dietro i firewall istituzionali e contemporaneamente utilizza il calcolo GPU se disponibile e esegue l’elaborazione batch. Rendiamo gratuitamente disponibile questo strumento ai ricercatori come alternativa alle costose opzioni a pagamento o alle opzioni gratuite meno efficaci attualmente disponibili.
Anche se la pulizia automatica dei dati è auspicabile, raramente uno sforzo di deidentificazione automatica è perfetto. Il rischio di perdita di IPF è di massima preoccupazione a causa delle conseguenze legali ed etiche. Incoraggiamo la comunità di ricerca a testare il protocollo sui rispettivi sistemi per la deidentificazione delle immagini. Il lavoro futuro include aggiornamenti al pacchetto software e l’incorporazione di feedback per renderlo più generalizzabile (inclusa la modifica dei formati di output) man mano che cresce l’adozione.
Per ulteriori informazioni sulle chiamate di funzione disponibili e altre documentazioni, leggere il paper qui.

Riferimenti
[1] E. Monteiro, C. Costa, J. L. Oliveira, Una pipeline di anonimizzazione per immagini mediche ecografiche in formato DICOM, Journal of medical systems, 41 (5) (2017) 1–16. [2] L. Fezai, T. Urruty, P. Bourdon, C. Fernandez-Maloigne, Anonimizzazione profonda delle immagini mediche, Multimedia Tools and Applications (2022) 1–15. [3] L.-C. Huang, H.-C. Chu, C.-Y. Lien, C.-H. Hsiao, T. Kao, Preservazione della privacy e protezione della sicurezza delle informazioni per i record sanitari elettronici portatili dei pazienti, Computers in Biology and Medicine 39 (9) (2009) 743–750. [4] D. Rodriguez Gonzalez, T. Carpenter, J. I. van Hemert, J. Wardlaw, Un toolkit open-source per l’anonimizzazione di immagini mediche, European radiology 20 (8) (2010) 1896–1904 [5] A. Kline, H. Wang, Y. Li, S. Dennis, M. Hutch, Z. Xu, F. Wang, F. Cheng, Y. Luo, Apprendimento automatico multimodale nella salute di precisione: una revisione della portata, npj Digital Medicine 5 (1) (2022) 1–14 [6] B. Liu, M. Zhu, Z. Zhang, C. Yin, Z. Liu, J. Gu, Conversione di immagini mediche con DICOM, in: 2007 Canadian Conference on Electrical and Computer Engineering, IEEE, 2007, pp. 36–39 [7] A. Kline, V. Appadurai, Y. Luo, S. Sanjiv, “Deanonimizzazione, pulizia e compressione di immagini mediche utilizzando Pylogik”, https://arxiv.org/abs/2304.12322
Inoltre, se ti piace leggere articoli come questo e desideri un accesso illimitato ai miei articoli e a tutti quelli forniti da Nisoo, considera di iscriverti utilizzando il mio link di riferimento qui sotto. La membership costa $5 (USD)/mese; guadagno una piccola commissione quando leggi i miei articoli che, a loro volta, contribuiscono a creare più contenuti e articoli!
Iscriviti a Nisoo con il mio link di riferimento — Adrienne Kline
Leggi ogni storia di Adrienne Kline (e di migliaia di altri scrittori su Nisoo). La tua quota di iscrizione supporta direttamente…
Nisoo.com



