V-Net, il fratello maggiore di U-Net nella segmentazione delle immagini
'V-Net, U-Net's older brother in image segmentation.
Benvenuti a questa guida sul V-Net, il cugino del ben noto U-Net, per la segmentazione di immagini 3D. Lo conoscerai a fondo!
Benvenuti in un emozionante viaggio nel mondo delle architetture di deep learning! Potresti già essere familiare con U-Net, un cambiamento di gioco nella computer vision che ha notevolmente ridisegnato il panorama della segmentazione delle immagini.
Oggi, mettiamo in evidenza il fratello maggiore di U-Net, il V-Net.
Pubblicato dai ricercatori Fausto Milletari, Nassir Navab e Seyed-Ahmad Ahmadi, l’articolo “VNet: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation” ha introdotto una metodologia innovativa per l’analisi di immagini 3D.
Questo articolo ti condurrà in un tour di questo articolo rivoluzionario, facendo luce sulle sue uniche contribuzioni e avanzamenti architettonici. Che tu sia un data scientist esperto, un appassionato di intelligenza artificiale in erba o semplicemente qualcuno interessato alle ultime tendenze tecnologiche, c’è qualcosa qui per te!
- Un’introduzione semplice al Bayesian Deep Learning
- Dimentica ChatGPT, questo nuovo assistente AI è anni luce avanti e cambierà per sempre il modo in cui lavori.
- Recensione di Synthesys Il miglior generatore di video AI? (Agosto 2023)
Un breve promemoria su U-Net
Prima di immergersi nel cuore di V-Net, prendiamoci un momento per apprezzare la sua ispirazione architettonica – U-Net. Non preoccuparti se questa è la tua prima introduzione a U-Net; ho tutto sotto controllo con un tutorial rapido e facile sull’architettura U-Net. È così conciso che capirai il concetto in non più di cinque minuti!
Ecco una breve panoramica di U-Net per un ripasso:

U-Net è famoso per la sua struttura simmetrica, che prende la forma di una ‘U’. Questa architettura è composta da due percorsi distinti:
- Percorso contrattivo (Sinistra): Qui riduciamo progressivamente la risoluzione dell’immagine aumentando il numero di filtri.
- Percorso espansivo (Destra): Questo percorso funge da immagine speculare del percorso contrattivo. Riduciamo gradualmente il numero di filtri aumentando la risoluzione fino a farla coincidere con la dimensione dell’immagine originale.
La bellezza di U-Net risiede nel suo uso innovativo di ‘connessioni residuali’ o ‘connessioni di salto’. Queste connettono strati corrispondenti nei percorsi contrattivo ed espansivo, consentendo alla rete di conservare dettagli ad alta risoluzione che vengono solitamente persi nel processo contrattivo.
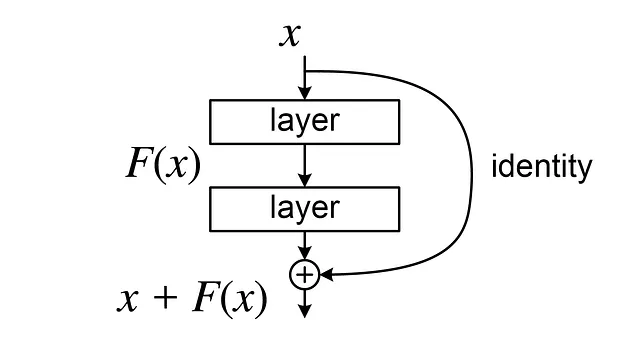
Perché è importante? Perché facilita il flusso del gradiente durante la retropropagazione, soprattutto nei primi strati. In sostanza, evitiamo il rischio di gradienti che tendono a zero, ostacolando il processo di apprendimento:
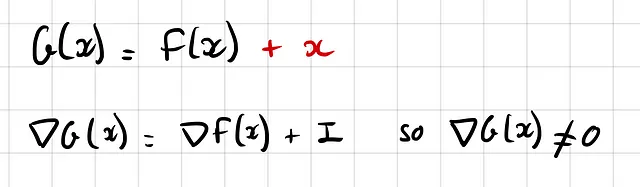
Ora, tenendo presente questa comprensione di U-Net, passiamo al mondo di V-Net. Nel suo cuore, V-Net condivide una filosofia simile di codificatore-decodificatore. Ma come scoprirai presto, ha un proprio insieme di caratteristiche uniche che lo distinguono dal suo fratello, U-Net.
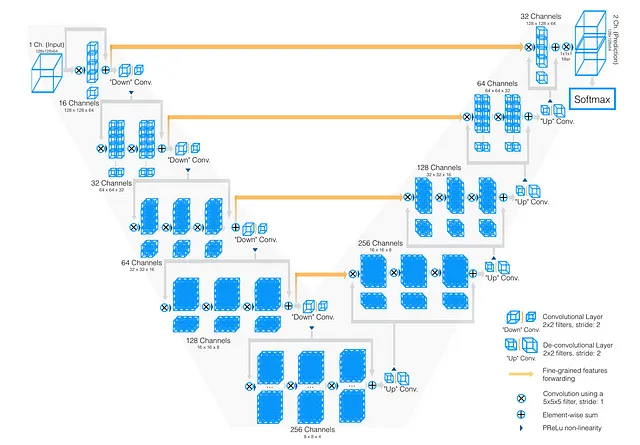
Cosa distingue V-Net da U-Net?
Andiamo avanti!
Differenza 1: Convoluzioni 3D invece di convoluzioni 2D
La prima differenza è chiara come il sole. Mentre U-Net è stato progettato per la segmentazione di immagini 2D, le immagini mediche spesso richiedono una prospettiva 3D (pensa alle scansioni volumetriche del cervello, alle scansioni TC, ecc.).
Ecco dove entra in gioco V-Net. La ‘V’ in V-Net sta per ‘Volumetrico’ e questo spostamento dimensionale richiede la sostituzione delle convoluzioni 2D con le convoluzioni 3D.
Differenza 2: Funzioni di attivazione, PreLU al posto di ReLU
Il campo dell’apprendimento profondo si è innamorato della funzione ReLU, grazie alla sua semplicità ed efficienza computazionale. Rispetto ad altre funzioni come sigmoide o tanh, ReLU è “non saturante”, il che significa che riduce il problema dei gradienti che svaniscono.
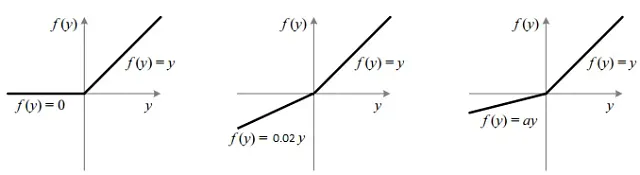
Ma ReLU non è perfetta. È notoriamente soggetta a un fenomeno noto come ‘Problema del ReLU che muore’, in cui molti neuroni producono sempre zero, diventando ‘neuroni morti’. Per contrastare questo, è stata introdotta LeakyReLU, che ha una pendenza piccola ma non nulla sul lato sinistro dello zero.
Portando il ragionamento ancora più avanti, V-Net sfrutta la Parametric ReLU (PReLU). Invece di codificare a mano la pendenza di LeakyReLU, perché non lasciare che la rete la impari?
Dopotutto, questa è una filosofia fondamentale dell’apprendimento profondo: vogliamo inserire il minor bias induttivo possibile e lasciare che il modello impari tutto da solo, supponendo di avere dati sufficienti.
Differenza 3: Diversa funzione di perdita basata sul punteggio di Dice
Ora, arriviamo forse al contributo più significativo di V-Net: un cambiamento nella funzione di perdita. A differenza della funzione di perdita con entropia incrociata di U-Net, V-Net utilizza la funzione di perdita di Dice.

Ma il problema principale di questa funzione è che non gestisce bene le classi sbilanciate. E questo problema è molto frequente nelle immagini mediche perché nella maggior parte dei casi lo sfondo è molto più presente rispetto alla zona di interesse.
Ad esempio, considera questa immagine:
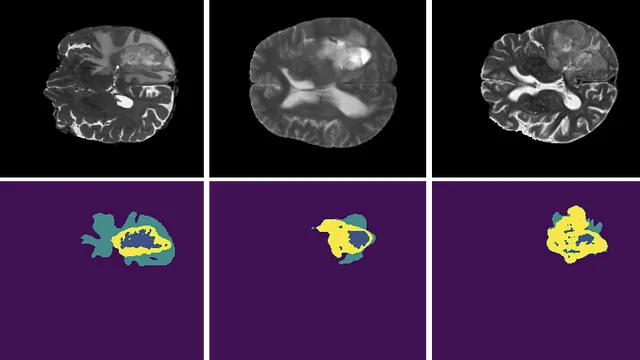
Come risultato, alcuni modelli possono diventare “pigri” e prevedere lo sfondo ovunque perché otterranno comunque una piccola perdita.
Quindi V-Net utilizza una funzione di perdita molto più efficace per questa questione: il coefficiente di Dice.
La ragione per cui è migliore è che misura l’overlap tra la zona prevista e la verità di riferimento come una proporzione, quindi la dimensione della classe viene presa in considerazione.
Anche se lo sfondo è quasi ovunque, il punteggio di Dice misura l’overlap tra la previsione e la verità di riferimento, quindi otteniamo comunque un numero compreso tra 0 e 1, anche se la classe è preponderante.
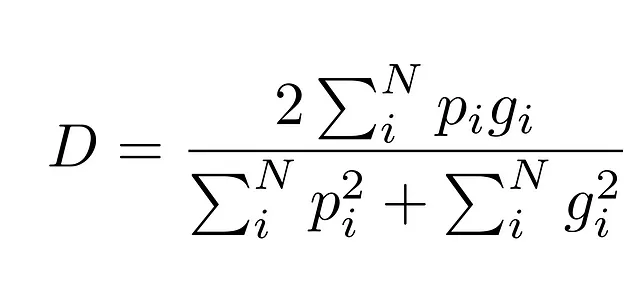
Dico che questo è forse il contributo principale dell’articolo perché passare dalle convoluzioni 2D alle convoluzioni 3D è un’idea molto naturale per gestire le immagini 3D. Tuttavia, questa funzione di perdita è stata ampiamente adottata nei compiti di segmentazione delle immagini.
Nella pratica, un approccio ibrido spesso si dimostra efficace, combinando la Cross Entropy Loss e la Dice Loss per sfruttare i punti di forza di entrambe.
Le prestazioni del V-Net
Quindi, abbiamo esplorato gli aspetti unici del V-Net, ma probabilmente ti stai chiedendo: “Tutta questa teoria è ottima, ma il V-Net funziona davvero nella pratica?” Bene, mettiamo alla prova il V-Net!
Gli autori hanno valutato le prestazioni del V-Net sul dataset PROMISE12.
Il dataset PROMISE12 è stato reso disponibile per la sfida di segmentazione della prostata MICCAI 2012.
Il V-Net è stato addestrato su 50 immagini di risonanza magnetica (MR), non moltissime!
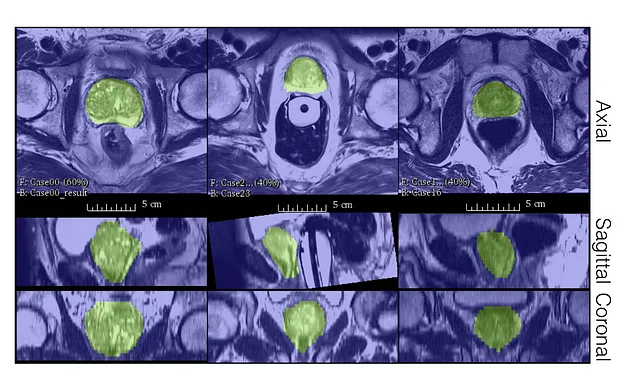
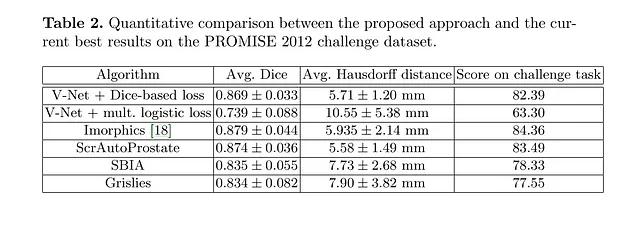
Come possiamo vedere, anche con poche etichette, il V-Net è in grado di produrre buone segmentazioni qualitative e ottenere un punteggio Dice molto buono.
Principali Limitazioni del V-Net
Effettivamente, il V-Net ha stabilito un nuovo punto di riferimento nel campo della segmentazione delle immagini, in particolare nell’imaging medico. Tuttavia, ogni innovazione ha spazio per crescere. Qui discuteremo alcune delle aree di rilievo in cui il V-Net potrebbe migliorare:
Limitazione 1: Dimensione del modello
Passare da 2D a 3D comporta un aumento significativo del consumo di memoria. Gli effetti a cascata di questo aumento sono molteplici:
- Il modello richiede uno spazio di memoria considerevole.
- Limita notevolmente la dimensione del lotto (poiché caricare più tensori 3D nella memoria GPU diventa impegnativo).
- I dati di imaging medico sono sparsi e costosi da etichettare, rendendo più difficile adattare un modello con così tanti parametri.
Limitazione 2: Non utilizza l’apprendimento non supervisionato o l’apprendimento auto-supervisionato
- Il V-Net opera esclusivamente in un contesto di apprendimento supervisionato, trascurando il potenziale dell’apprendimento non supervisionato. In un campo in cui le scansioni non etichettate superano significativamente quelle annotate, l’incorporazione dell’apprendimento non supervisionato potrebbe essere rivoluzionaria.
Limitazione 3: Nessuna stima dell’incertezza
- Il V-Net non stima le incertezze, il che significa che non può valutare la propria fiducia nelle sue previsioni. Questo è un ambito in cui il Bayesian Deep Learning brilla. (Fai riferimento a questo post per una Gentile Introduzione al Bayesian Deep Learning).
Limitazione 4: Mancanza di Robustezza
- Le reti neurali convoluzionali (CNN) tradizionalmente hanno difficoltà con la generalizzazione. Non sono robuste rispetto a variazioni come il cambio di contrasto, le distribuzioni multimodali o le diverse risoluzioni. Anche qui il V-Net potrebbe migliorare.
Conclusione
Il V-Net, il meno conosciuto ma potente controparte dell’U-Net, ha rivoluzionato la computer vision, in particolare la segmentazione delle immagini. La sua transizione da immagini 2D a 3D e l’introduzione del coefficiente Dice, ora uno strumento ubiquo, hanno stabilito nuovi standard nel campo.
Nonostante le sue limitazioni, il V-Net dovrebbe essere il modello di riferimento per chiunque affronti un compito di segmentazione di immagini 3D. Per ulteriori miglioramenti, esplorare l’apprendimento non supervisionato e integrare meccanismi di attenzione sembra essere un’ottima strada da percorrere.
Se hai feedback, idee da condividere, vuoi lavorare con me o semplicemente vuoi salutare, compila il modulo qui sotto e iniziamo una conversazione.
Dì Ciao 🌿
Non esitare a lasciare un applauso o a seguirmi per altri contenuti!
Riferimenti
- Cucinare il tuo primo U-Net per la segmentazione delle immagini
- Un’introduzione alle basi del Bayesian Deep Learning
- Milletari, F., Navab, N., & Ahmadi, S. A. (2016). V-Net: Reti Neurali completamente convoluzionali per la segmentazione di immagini mediche volumetriche. In 3D Vision (3DV), 2016 Quarta Conferenza Internazionale su (pp. 565–571). IEEE.
- Ronneberger, O., Fischer, P., & Brox, T. (2015). U-Net: Reti Convoluzionali per la Segmentazione Biomedica delle Immagini. In Conferenza Internazionale su Elaborazione delle Immagini Mediche e Intervento Assistito dal Computer (pp. 234–241). Springer, Cham.





