La guida definitiva a nnU-Net
Guida a nnU-Net
Tutto quello che devi sapere per capire lo Stato dell’Arte di nnU-Net e come applicarlo ai tuoi dati.

Durante il mio stage di ricerca in Deep Learning e Neuroscienze presso l’Università di Cambridge, ho utilizzato molto nnU-Net, che è una solida base nel campo della Segmentazione Semantica delle Immagini.
Tuttavia, ho avuto qualche difficoltà a comprendere appieno il modello e come allenarlo, e non ho trovato molta assistenza su internet. Ora che mi sento a mio agio, ho creato questo tutorial per aiutarti, sia nella tua ricerca di una migliore comprensione di ciò che si cela dietro questo modello, sia nell’applicarlo ai tuoi stessi dati.
In questa guida imparerai:
- Sviluppare una panoramica concisa dei principali contributi di nnU-Net.
- Come applicare nnU-Net ai tuoi dati.
Tutto il codice è disponibile in questo notebook di Google Collab.
- 7 Lezioni apprese sulla creazione di un prodotto completo utilizzando ChatGPT
- Valutazione dinamica del progetto di intelligenza artificiale
- Proteggi la tua app LLM. Un must da leggere!
Questo lavoro mi ha richiesto molto tempo e sforzo. Se trovi utile questo contenuto, ti preghiamo di considerare di seguirmi per aumentarne la visibilità e aiutare a creare altri tutorial simili!
Una Breve Storia di nnU-Net
Riconosciuto come un modello all’avanguardia nella Segmentazione delle Immagini, nnU-Net rappresenta una forza indomabile sia nel processing di immagini 2D che 3D. Le sue prestazioni sono così robuste che rappresenta una base solida con cui nuove architetture di computer vision vengono confrontate. In sostanza, se ti stai avventurando nel mondo dello sviluppo di modelli di visione artificiale innovativi, considera nnU-Net come il tuo “obiettivo da superare”.
Questo potente strumento si basa sul modello U-Net (puoi trovare uno dei miei tutorial qui: Crea il tuo primo U-Net), che ha fatto il suo debutto nel 2015. L’appellativo “nnU-Net” sta per “No New U-Net”, un omaggio al fatto che il suo design non introduce modifiche architettoniche rivoluzionarie. Al contrario, prende la struttura U-Net esistente e ne sfrutta appieno il potenziale attraverso un insieme di strategie di ottimizzazione ingenue.
A differenza di molti moderni reti neurali, nnU-Net non si basa su connessioni residue, connessioni dense o meccanismi di attenzione. La sua forza risiede nella sua meticolosa strategia di ottimizzazione, che include tecniche come il campionamento, la normalizzazione, la scelta oculata della funzione di perdita, le impostazioni dell’ottimizzatore, l’aumento dei dati, l’inferenza basata su patch e l’ensemble tra i modelli. Questo approccio olistico consente a nnU-Net di spingere i limiti di ciò che è possibile ottenere con l’architettura originale di U-Net.
Esplorazione di Architetture Diverse all’interno di nnU-Net
Anche se potrebbe sembrare un’entità unica, nnU-Net è in realtà un termine ombrello per tre tipi distinti di U-Net:
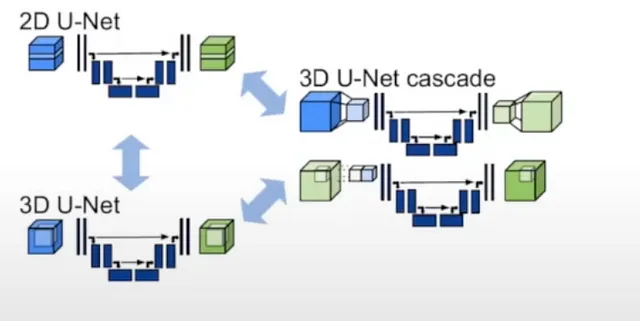
- U-Net 2D: Probabilmente la variante più conosciuta, opera direttamente su immagini 2D.
- U-Net 3D: Questa è un’estensione di U-Net 2D in grado di gestire direttamente immagini 3D tramite l’applicazione di convoluzioni 3D.
- U-Net Cascade: Questo modello genera segmentazioni a bassa risoluzione e successivamente le perfeziona.
Ognuna di queste architetture porta i suoi punti di forza unici sul tavolo e, inevitabilmente, ha determinate limitazioni.
Ad esempio, impiegare un 2D U-Net per la segmentazione di immagini 3D potrebbe sembrare controintuitivo, ma nella pratica può comunque essere altamente efficace. Ciò viene ottenuto suddividendo il volume 3D in piani 2D.
Sebbene un 3D U-Net possa sembrare più sofisticato, dato il suo numero di parametri più elevato, non è sempre la soluzione più efficiente. In particolare, i 3D U-Net spesso incontrano difficoltà con l’anisotropia, che si verifica quando le risoluzioni spaziali differiscono lungo diversi assi (ad esempio, 1 mm lungo l’asse x e 1,2 mm lungo l’asse z).
La variante U-Net Cascade diventa particolarmente utile quando si lavora con dimensioni di immagini grandi. Essa impiega un modello preliminare per condensare l’immagine, seguito da un 3D U-Net standard che restituisce segmentazioni a bassa risoluzione. Le predizioni generate vengono quindi ingrandite, ottenendo un’output raffinato e completo.
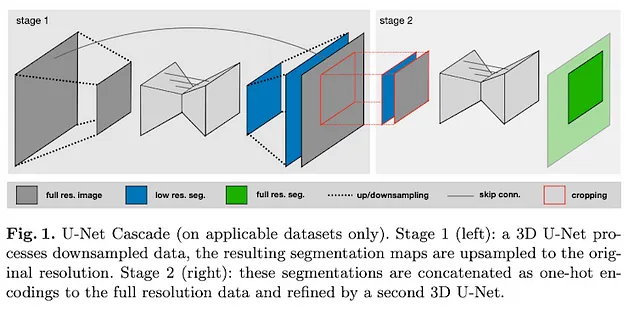
Di solito, la metodologia prevede di allenare tutte e tre le varianti del modello all’interno del framework nnU-Net. Il passaggio successivo potrebbe essere quello di scegliere il migliore tra i tre o impiegare tecniche di ensemble. Una di queste tecniche potrebbe consistere nell’integrare le predizioni sia dei 2D U-Net che dei 3D U-Net.
Tuttavia, è importante notare che questa procedura può richiedere molto tempo (e anche denaro perché sono necessari crediti GPU). Se i vincoli consentono solo l’allenamento di un singolo modello, non preoccuparti. Puoi scegliere di allenare solo un modello, poiché il modello di ensemble apporta solo guadagni molto marginali.
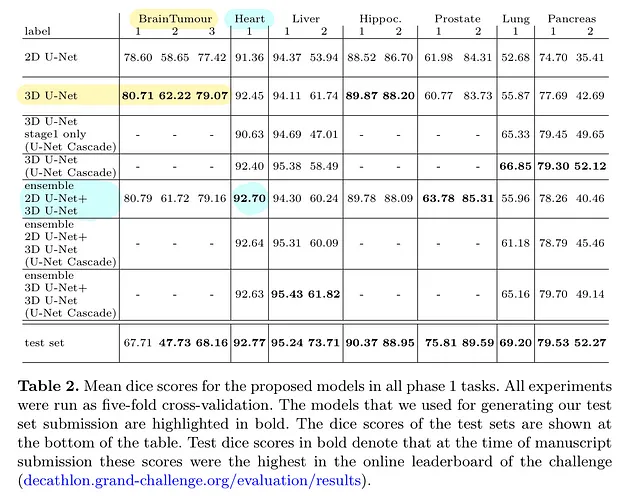
Questa tabella illustra la variante di modello con le migliori prestazioni in relazione a dataset specifici:
Adattamento dinamico delle topologie di rete
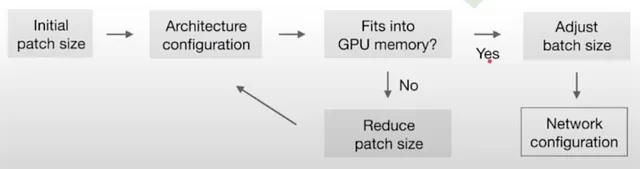
Dati i significativi scostamenti nelle dimensioni delle immagini (considera la forma mediana di 482 × 512 × 512 per le immagini del fegato rispetto a 36 × 50 × 35 per le immagini dell’ippocampo), nnU-Net adatta intelligentemente la dimensione del patch di input e il numero di operazioni di pooling per ogni asse. Ciò implica essenzialmente un adattamento automatico del numero di strati convoluzionali per ogni dataset, facilitando l’aggregazione efficace delle informazioni spaziali. Oltre all’adattamento alle geometrie delle immagini variegate, questo modello tiene conto di vincoli tecnici, come la memoria disponibile.
È fondamentale notare che il modello non esegue la segmentazione direttamente sull’intera immagine, ma invece su patch accuratamente estratte con regioni sovrapposte. Le predizioni su queste patch vengono successivamente mediate, portando all’output finale della segmentazione.
Tuttavia, avere una patch grande significa un maggiore utilizzo della memoria, e anche la dimensione del batch consuma memoria. Il compromesso adottato è sempre quello di dare priorità alla dimensione della patch (capacità del modello) piuttosto che alla dimensione del batch (utile solo per l’ottimizzazione).
Ecco l’algoritmo euristico utilizzato per calcolare la dimensione ottimale della patch e del batch:
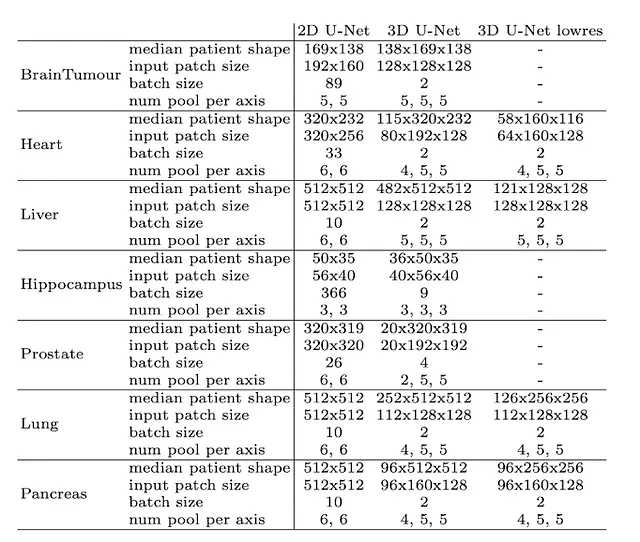
Ecco come appare per diversi dataset e dimensioni di input:
Ottimo! Ora passiamo rapidamente in rassegna tutte le tecniche utilizzate in nnU-Net:
Allenamento
Tutti i modelli vengono allenati da zero e valutati utilizzando una cross-validation a cinque fold sul set di allenamento, il che significa che il set di allenamento originale viene diviso in modo casuale in cinque parti uguali, o ‘fold’. In questo processo di cross-validation, quattro di queste fold vengono utilizzate per l’allenamento del modello, mentre la restante fold viene utilizzata per la valutazione o il testing. Questo processo viene quindi ripetuto cinque volte, con ciascuna delle cinque fold utilizzata esattamente una volta come set di valutazione.
Per la perdita, utilizziamo una combinazione di Dice e Cross Entropy Loss. Questa è una perdita molto frequente nella segmentazione delle immagini. Ulteriori dettagli sulla Dice Loss in V-Net, il fratello maggiore di U-Net
Tecniche di aumento dei dati
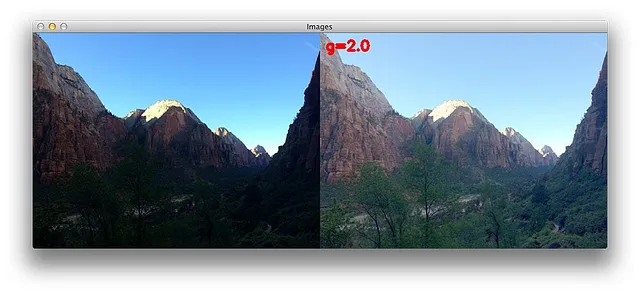
L’nnU-Net ha un robusto pipeline di aumento dei dati. Gli autori utilizzano rotazioni casuali, ridimensionamenti casuali, deformazioni elastiche casuali, correzione gamma e specchiatura.
NB: Puoi aggiungere le tue trasformazioni modificando il codice sorgente

Inferenza basata su patch
Quindi, come abbiamo detto, il modello non prevede direttamente sull’immagine a piena risoluzione, ma lo fa su patch estratte e quindi aggrega la previsione.
Ecco come appare:
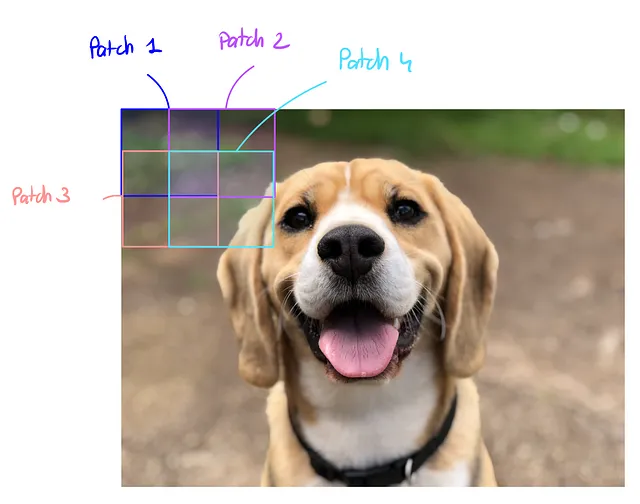
NB: Le patch al centro dell’immagine hanno un peso maggiore rispetto a quelle ai lati, perché contengono più informazioni e il modello funziona meglio su di esse
Accoppiamento dei modelli a coppie
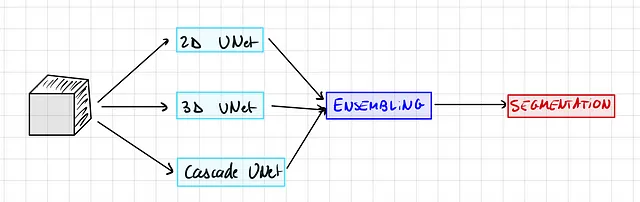
Quindi, se ricordate bene, possiamo addestrare fino a 3 modelli diversi, 2D, 3D e a cascata. Ma quando facciamo inferenza possiamo utilizzare solo un modello alla volta, giusto?
Bene, si scopre che no, modelli diversi hanno punti di forza e debolezza diversi. Quindi possiamo effettivamente combinare le previsioni di diversi modelli in modo che se un modello è molto fiducioso, diamo priorità alla sua previsione.
nnU-Net testa ogni combinazione di 2 modelli tra i 3 modelli disponibili e sceglie il migliore.
Nella pratica, ci sono 2 modi per farlo:
Voto duro: Per ogni pixel, guardiamo tutte le probabilità generate dai 2 modelli e prendiamo la classe con la probabilità più alta.
Voto soft: Per ogni pixel, facciamo la media delle probabilità dei modelli e poi prendiamo la classe con la massima probabilità.
Implementazione pratica
Prima di iniziare, puoi scaricare il dataset qui e seguire il notebook di Google Collab.
Se non hai capito nulla della prima parte, non preoccuparti, questa è la parte pratica, devi solo seguirmi e otterrai comunque i migliori risultati.
Hai bisogno di una GPU per addestrare il modello, altrimenti non funziona. Puoi farlo localmente o su Google Collab, non dimenticare di cambiare il runtime > GPU
Quindi, prima di tutto, devi avere un dataset pronto con immagini di input e la relativa segmentazione. Puoi seguire il mio tutorial scaricando questo dataset pronto per la segmentazione cerebrale 3D e poi puoi sostituirlo con il tuo dataset.
Download dei dati
Prima di tutto, dovresti scaricare i tuoi dati e posizionarli nella cartella dei dati, denominando le due cartelle “input” e “ground_truth” che contiene la segmentazione.
Per il resto del tutorial userò il dataset MindBoggle per la segmentazione delle immagini. Puoi scaricarlo su questo Google Drive:
Ci vengono fornite scansioni MRI 3D del cervello e vogliamo segmentare la materia bianca e grigia:
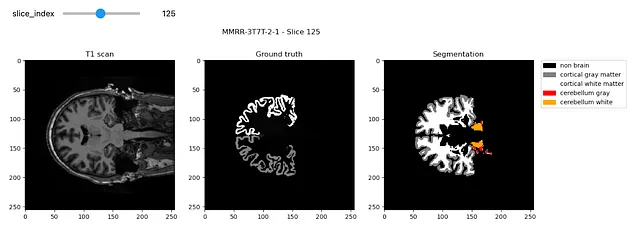
Dovrebbe apparire così:
Impostazione della directory principale
Se esegui questo su Google Colab, imposta collab = True, altrimenti collab = False
collab = Trueimport osimport shutil#bibliotechefrom collections import OrderedDictimport jsonimport numpy as np#visualizzazione del datasetimport matplotlib.pyplot as pltimport nibabel as nibif collab: from google.colab import drive drive.flush_and_unmount() drive.mount('/content/drive', force_remount=True) # Cambia "neurosciences-segmentation" con il nome della tua cartella di progetto root_dir = "/content/drive/MyDrive/neurosciences-segmentation"else: # ottieni la directory della directory genitore root_dir = os.getcwd()input_dir = os.path.join(root_dir, 'data/input')segmentation_dir = os.path.join(root_dir, 'data/ground_truth')my_nnunet_dir = os.path.join(root_dir,'my_nnunet')print(my_nnunet_dir)Ora stiamo per definire una funzione che crea le cartelle per noi:
def make_if_dont_exist(folder_path,overwrite=False): """ crea una cartella se non esiste input: folder_path : percorso relativo della cartella che deve essere creata over_write :(default: False) se True sovrascrive la cartella esistente """ if os.path.exists(folder_path): if not overwrite: print(f'{folder_path} esiste.') else: print(f"{folder_path} sovrascritto") shutil.rmtree(folder_path) os.makedirs(folder_path) else: os.makedirs(folder_path) print(f"{folder_path} creato!")E utilizziamo questa funzione per creare la nostra cartella “my_nnunet” in cui verrà salvato tutto
os.chdir(root_dir)make_if_dont_exist('my_nnunet', overwrite=False)os.chdir('my_nnunet')print(f"Directory di lavoro corrente: {os.getcwd()}")Installazione delle librerie
Ora stiamo per installare tutti i requisiti. Prima installiamo la libreria nnunet. Se sei in un notebook, esegui questo in una cella:
!pip install nnunetAltrimenti puoi installare nnunet direttamente dal terminale con
pip install nnunetOra stiamo per clonare il repository git di nnUnet e NVIDIA apex. Questo contiene gli script di addestramento così come un acceleratore GPU.
!git clone https://github.com/MIC-DKFZ/nnUNet.git!git clone https://github.com/NVIDIA/apex# repository dir è il percorso della cartella githubrespository_dir = os.path.join(my_nnunet_dir,'nnUNet')os.chdir(respository_dir)!pip install -e!pip install --upgrade git+https://github.com/nanohanno/hiddenlayer.git@bugfix/get_trace_graph#egg=hiddenlayerCreazione delle cartelle
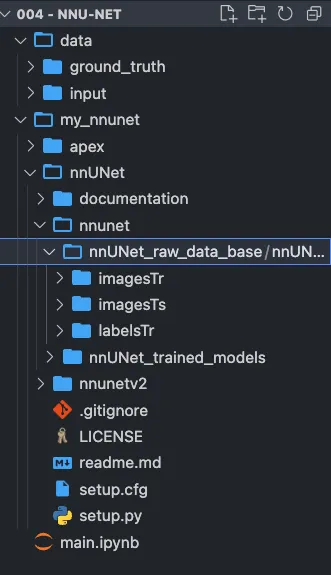
nnUnet richiede una struttura molto specifica per le cartelle.
task_name = 'Task001' #cambia qui per un nome di attività diverso# Definiamo tutti i percorsi necessarinnunet_dir = "nnUNet/nnunet/nnUNet_raw_data_base/nnUNet_raw_data"task_folder_name = os.path.join(nnunet_dir,task_name) train_image_dir = os.path.join(task_folder_name,'imagesTr') # percorso delle immagini di addestramentotrain_label_dir = os.path.join(task_folder_name,'labelsTr') # percorso delle etichette di addestramentotest_dir = os.path.join(task_folder_name,'imagesTs') # percorso delle immagini di testmain_dir = os.path.join(my_nnunet_dir,'nnUNet/nnunet') # percorso della directory principaletrained_model_dir = os.path.join(main_dir, 'nnUNet_trained_models') # percorso dei modelli addestratiOriginariamente, nnU-Net è stato progettato per una sfida decathlon con compiti diversi. Se hai compiti diversi, esegui questa cella per tutti i tuoi compiti.
# Creazione di tutte le cartelleoverwrite = False # Imposta su True se desideri sovrascrivere le cartellemake_if_dont_exist(task_folder_name,overwrite = overwrite)make_if_dont_exist(train_image_dir, overwrite = overwrite)make_if_dont_exist(train_label_dir, overwrite = overwrite)make_if_dont_exist(test_dir,overwrite= overwrite)make_if_dont_exist(trained_model_dir, overwrite=overwrite)Dovresti avere una struttura del genere:
Impostazione delle variabili di ambiente
Lo script deve sapere dove hai inserito i tuoi dati grezzi, dove può trovare i dati preelaborati e dove deve salvare i risultati.
os.environ['nnUNet_raw_data_base'] = os.path.join(main_dir,'nnUNet_raw_data_base')os.environ['nnUNet_preprocessed'] = os.path.join(main_dir,'preprocessed')os.environ['RESULTS_FOLDER'] = trained_model_dirSposta i file nei repository corretti:
Definiamo una funzione che sposterà le nostre immagini nei repository corretti nella cartella nnunet:
def copy_and_rename(old_location,old_file_name,new_location,new_filename,delete_original = False): shutil.copy(os.path.join(old_location,old_file_name),new_location) os.rename(os.path.join(new_location,old_file_name),os.path.join(new_location,new_filename)) if delete_original: os.remove(os.path.join(old_location,old_file_name))Ora eseguiamo questa funzione per le immagini di input e di ground truth:
list_of_all_files = os.listdir(segmentation_dir)list_of_all_files = [file_name for file_name in list_of_all_files if file_name.endswith('.nii.gz')]for file_name in list_of_all_files: copy_and_rename(input_dir,file_name,train_image_dir,file_name) copy_and_rename(segmentation_dir,file_name,train_label_dir,file_name)Ora dobbiamo rinominare i file per essere accettati dal formato nnUnet, ad esempio subject.nii.gz diventerà subject_0000.nii.gz
def check_modality(filename): """ Verifica l'esistenza della modalità restituisce False se la modalità non viene trovata altrimenti True """ end = filename.find('.nii.gz') modality = filename[end-4:end] for mod in modality: if not(ord(mod)>=48 and ord(mod)<=57): #se non è un numero tra 0 e 9 return False return Truedef rename_for_single_modality(directory): for file in os.listdir(directory): if check_modality(file)==False: new_name = file[:file.find('.nii.gz')]+"_0000.nii.gz" os.rename(os.path.join(directory,file),os.path.join(directory,new_name)) print(f"Rinominato in {new_name}") else: print(f"Modalità presente: {file}")rename_for_single_modality(train_image_dir)# rename_for_single_modality(test_dir)Configurazione del file JSON
Siamo quasi finiti!
Dovresti modificare principalmente 2 cose:
- La modalità (se è CT o MRI, ciò cambia la normalizzazione)
- Le etichette: Inserisci le tue classi
overwrite_json_file = True # Impostalo su True se vuoi sovrascrivere il file dataset.json nella cartella Taskjson_file_exist = Falseif os.path.exists(os.path.join(task_folder_name,'dataset.json')): print('dataset.json esiste già!') json_file_exist = Trueif json_file_exist==False or overwrite_json_file: json_dict = OrderedDict() json_dict['name'] = task_name json_dict['description'] = "Segmentazione di scansioni T1 da MindBoggle" json_dict['tensorImageSize'] = "3D" json_dict['reference'] = "vedi il sito web della sfida" json_dict['licence'] = "vedi il sito web della sfida" json_dict['release'] = "0.0" ######################## MODIFICA QUESTO ######################## #puoi menzionare più di una modalità json_dict['modality'] = { "0": "MRI" } #le etichette+1 devono essere menzionate per tutte le etichette nel dataset json_dict['labels'] = { "0": "Non cerebrale", "1": "Materia grigia corticale", "2": "Materia bianca corticale", "3" : "Materia grigia del cervelletto", "4" : "Materia bianca del cervelletto" } ############################################################# train_ids = os.listdir(train_label_dir) test_ids = os.listdir(test_dir) json_dict['numTraining'] = len(train_ids) json_dict['numTest'] = len(test_ids) #nessuna modalità nell'immagine di train e nelle etichette nel dataset.json json_dict['training'] = [{'image': "./imagesTr/%s" % i, "label": "./labelsTr/%s" % i} for i in train_ids] #rimuovere la modalità dal nome dell'immagine di test per essere salvata in dataset.json json_dict['test'] = ["./imagesTs/%s" % (i[:i.find("_0000")]+'.nii.gz') for i in test_ids] with open(os.path.join(task_folder_name,"dataset.json"), 'w') as f: json.dump(json_dict, f, indent=4, sort_keys=True) if os.path.exists(os.path.join(task_folder_name,'dataset.json')): if json_file_exist==False: print('dataset.json creato!') else: print('dataset.json sovrascritto!')Preelaborazione dei dati per il formato nnU-Net
Questo crea il dataset per il formato nnU-Net
# -t 1 significa "Task001", se hai un task diverso cambialo!nnUNet_plan_and_preprocess -t 1 --verify_dataset_integrityAllenamento dei modelli
Siamo ora pronti per allenare i modelli!
Per allenare il 3D U-Net:
#allenare il 3D full resolution U net!nnUNet_train 3d_fullres nnUNetTrainerV2 1 0 --npz Per allenare il 2D U-Net:
# allenare il 2D U net!nnUNet_train 2d nnUNetTrainerV2 1 0 --npzPer allenare il modello a cascata:
# allenare il 3D U-net a cascata!nnUNet_train 3d_lowres nnUNetTrainerV2CascadeFullRes 1 0 --npz!nnUNet_train 3d_fullres nnUNetTrainerV2CascadeFullRes 1 0 --npzNota: Se metti in pausa l’allenamento e vuoi riprenderlo, aggiungi un “-c” alla fine per “continuare”.
Per esempio:
#allenare il 3D full resolution U net!nnUNet_train 3d_fullres nnUNetTrainerV2 1 0 --npz Inferenza
Ora possiamo eseguire l’inferenza:
result_dir = os.path.join(task_folder_name, 'nnUNet_Prediction_Results')make_if_dont_exist(result_dir, overwrite=True)# -i è la cartella di input# -o è dove vuoi salvare le previsioni# -t 1 significa task 1, cambialo se hai un numero di task diverso# Usa -m 2d, o -m 3d_fullres, o -m 3d_cascade_fullres!nnUNet_predict -i /content/drive/MyDrive/neurosciences-segmentation/my_nnunet/nnUNet/nnunet/nnUNet_raw_data_base/nnUNet_raw_data/Task001/imagesTs -o /content/drive/MyDrive/neurosciences-segmentation/my_nnunet/nnUNet/nnunet/nnUNet_raw_data_base/nnUNet_raw_data/Task001/nnUNet_Prediction_Results -t 1 -tr nnUNetTrainerV2 -m 2d -f 0 --num_threads_preprocessing 1Visualizzazione delle previsioni
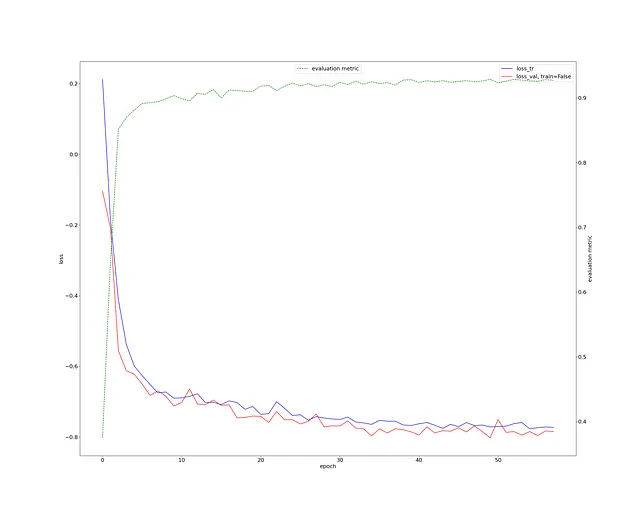
Prima controlliamo la perdita di allenamento. Questo sembra molto sano e abbiamo un punteggio Dice > 0.9 (curva verde).
Questo è davvero eccellente per così poco lavoro e un compito di segmentazione neuroimaging 3D.
Guardiamo un campione:
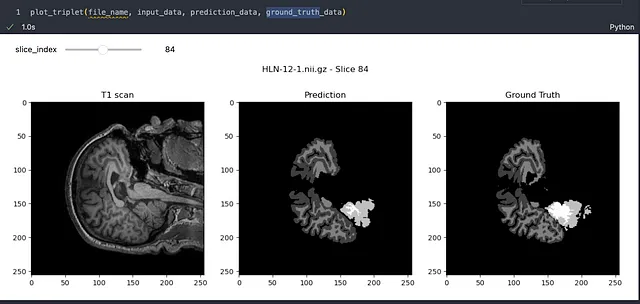
I risultati sono davvero impressionanti! È chiaro che il modello ha imparato efficacemente come segmentare le immagini cerebrali con grande precisione. Anche se possono esserci imperfezioni minori, è importante ricordare che il campo della segmentazione delle immagini sta avanzando rapidamente e stiamo compiendo progressi significativi verso la perfezione.
In futuro, c’è spazio per ottimizzare ulteriormente le prestazioni di nnU-Net, ma questo sarà oggetto di un altro articolo
Se hai trovato questo articolo interessante e utile, considera di seguirmi per approfondimenti nel mondo dell’apprendimento profondo. Il tuo supporto mi aiuta a continuare a produrre contenuti che contribuiscono alla nostra comprensione collettiva.
Se hai feedback, idee da condividere, vuoi lavorare con me o semplicemente vuoi salutare, compila il modulo qui sotto e iniziamo una conversazione.
Dì Ciao 🌿
Non esitare a lasciare un applauso o a seguirmi per altri contenuti!
Riferimenti
- Ronneberger, O., Fischer, P., & Brox, T. (2015). U-net: Reti convoluzionali per la segmentazione di immagini biomediche. In Conferenza internazionale sull’elaborazione di immagini mediche e l’intervento assistito dal computer (pp. 234-241). Springer, Cham.
- Isensee, F., Jaeger, P. F., Kohl, S. A., Petersen, J., & Maier-Hein, K. H. (2021). nnU-Net: un metodo di auto-configurazione per la segmentazione di immagini biomediche basato sull’apprendimento profondo. Nature Methods, 18(2), 203-211.
- Ioffe, S., & Szegedy, C. (2015). Batch normalization: Accelerazione dell’addestramento di reti neurali profonde mediante la riduzione dello shift covariato interno. Articolo preprint di arXiv arXiv:1502.03167.
- Ulyanov, D., Vedaldi, A., & Lempitsky, V. (2016). Normalizzazione delle istanze: l’ingrediente mancante per la stilizzazione veloce. Articolo preprint di arXiv arXiv:1607.08022.
- Dataset MindBoggle