Utilizzo delle Reti Bayesiane per prevedere il volume dei servizi ausiliari negli ospedali
Utilizzo Reti Bayesiane per prevedere volume servizi ausiliari ospedali
Un esempio Python che utilizza variabili di input diagnostiche

Dal momento che lavoro con dati sanitari (ormai da quasi 10 anni), prevedere il volume futuro dei pazienti è stato un problema difficile da risolvere. Ci sono così tante dipendenze da considerare: richieste e gravità dei pazienti, esigenze amministrative, vincoli delle stanze per gli esami, un medico che ha appena chiamato per dire che è malato, una tempesta di neve improvvisa. Inoltre, scenari imprevisti possono avere un impatto a cascata sulla pianificazione e l’allocazione delle risorse che contraddicono anche le migliori previsioni in Excel.
Questi tipi di problemi sono davvero interessanti da cercare di risolvere dal punto di vista dei dati, sia perché sono difficili e ci si può dedicare per un po’ di tempo, sia perché anche piccoli miglioramenti possono portare a grandi successi (ad esempio, migliorare il flusso dei pazienti, ridurre i tempi di attesa, rendere i medici più felici, ridurre i costi).
Come risolverlo allora? Beh, Epic ci fornisce molti dati, compresi i record effettivi di quando i pazienti arrivano per le loro visite. Con le uscite storiche note, ci troviamo principalmente nello spazio dell’apprendimento supervisionato, e le Reti Bayesiane (BN) sono buoni modelli grafici probabilistici.
Mentre la maggior parte delle decisioni può essere presa su un singolo input (ad esempio, “dovrei portare un impermeabile?”, se l’input è “sta piovendo”, allora la decisione è “sì”), le BN possono gestire facilmente decisioni più complesse, che coinvolgono input multipli, di probabilità e dipendenze variabili. In questo articolo, voglio “scarabocchiare” in python una BN super semplice che può restituire un punteggio di probabilità per un paziente che arriva tra 2 mesi, basato su probabilità note per tre fattori: sintomi, stadio del cancro e obiettivo di trattamento.
- È difficile la Data Science? Conosci la realtà
- Google AI presenta Visually Rich Document Understanding (VRDU) un dataset per il miglioramento del monitoraggio del progresso del compito di comprensione dei documenti.
- Possono i modelli di linguaggio su larga scala sostituire gli esseri umani nelle attività di valutazione del testo? Questo articolo sull’IA propone di utilizzare i modelli di linguaggio su larga scala per valutare la qualità dei testi come alternativa alla valutazione umana.
Comprendere le Reti Bayesiane:
Alla base, una Rete Bayesiana è una rappresentazione grafica di una distribuzione di probabilità congiunta utilizzando un grafo aciclico diretto (DAG). I nodi nel DAG rappresentano variabili casuali, e gli archi diretti indicano relazioni causali o dipendenze condizionali tra queste variabili. Come accade per tutti i progetti di data science, passare molto tempo con gli stakeholder all’inizio per mappare correttamente i flussi di lavoro (ad esempio, le variabili) coinvolti nella presa di decisioni è fondamentale per ottenere previsioni di alta qualità.
Quindi, inventerò uno scenario in cui incontriamo i nostri partner di oncologia del seno e spiegheranno che tre variabili sono fondamentali per determinare se un paziente avrà bisogno di un appuntamento tra 2 mesi: i loro sintomi, lo stadio del cancro e l’obiettivo di trattamento. Sto inventando mentre scrivo, ma andiamo avanti.
(In realtà ci saranno decine di fattori che influenzano i volumi futuri dei pazienti, alcuni con dipendenze singole o multiple, altri completamente indipendenti ma che influenzano comunque).
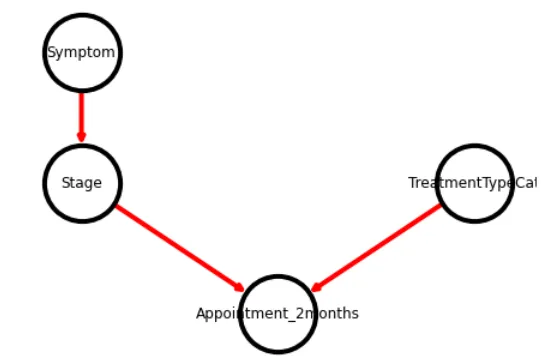
Direi che il flusso di lavoro assomiglia a quello sopra: lo stadio dipende dai sintomi, ma il tipo di trattamento è indipendente da quelli e influenza anche l’appuntamento tra 2 mesi.
Basandoci su questo, recupereremmo i dati per queste variabili dalla nostra fonte di dati (per noi, Epic), che ancora una volta conterrà valori noti per il nostro nodo di punteggio (Appointment_2months), etichettati come “sì” o “no”.
# install the packagesimport pandas as pd # per la manipolazione dei dati import networkx as nx # per disegnare i grafiimport matplotlib.pyplot as plt # per disegnare i grafi!pip install pybbn# per creare le Reti Bayesiane di Credenze (BBN)from pybbn.graph.dag import Bbnfrom pybbn.graph.edge import Edge, EdgeTypefrom pybbn.graph.jointree import EvidenceBuilderfrom pybbn.graph.node import BbnNodefrom pybbn.graph.variable import Variablefrom pybbn.pptc.inferencecontroller import InferenceController# Creare i nodi digitando manualmente le probabilitàSymptom = BbnNode(Variable(0, 'Sintomo', ['Non-Maligno', 'Maligno']), [0.30658, 0.69342])Stage = BbnNode(Variable(1, 'Stadio', ['Stadio_III_IV', 'Stadio_I_II']), [0.92827, 0.07173, 0.55760, 0.44240])TreatmentTypeCat = BbnNode(Variable(2, 'CategoriaTipoTrattamento', ['Adiuvante/Neoadiuvante', 'Trattamento', 'Terapia']), [0.58660, 0.24040, 0.17300])Appointment_2weeks = BbnNode(Variable(3, 'Appuntamento_2settimane', ['No', 'Sì']), [0.92314, 0.07686, 0.89072, 0.10928, 0.76008, 0.23992, 0.64250, 0.35750, 0.49168, 0.50832, 0.32182, 0.67818])In alto, immettiamo manualmente alcuni punteggi di probabilità per i livelli in ogni variabile (nodo). Nella pratica, si utilizzerebbe una tabella incrociata per ottenere questo.
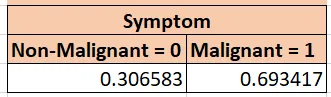
Ad esempio, per la variabile sintomo, otterrò le frequenze dei suoi 2 livelli, circa il 31% sono non maligni e il 69% sono maligni.
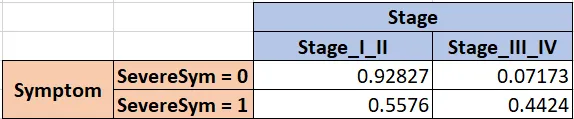
Quindi, consideriamo la variabile successiva, Fase, e incrociamo questa con il Sintomo per ottenere queste frequenze.
E così via, fino a quando tutte le tabelle incrociate tra le coppie genitore-figlio sono definite.
Ora, la maggior parte delle BN include molte relazioni genitore-figlio, quindi calcolare le probabilità può diventare noioso (e soprattutto soggetto a errori), quindi la funzione sottostante può calcolare la matrice delle probabilità per qualsiasi nodo figlio corrispondente a 0, 1 o 2 genitori.
# Questa funzione aiuta a calcolare la distribuzione di probabilità, che viene inserita in BBN (nota, può gestire fino a 2 genitori)def probs(data, child, parent1=None, parent2=None): if parent1==None: # Calcola le probabilità prob=pd.crosstab(data[child], 'Vuoto', margins=False, normalize='columns').sort_index().to_numpy().reshape(-1).tolist() elif parent1!=None: # Verifica se il nodo figlio ha 1 genitore o 2 genitori if parent2==None: # Calcola le probabilità prob=pd.crosstab(data[parent1],data[child], margins=False, normalize='index').sort_index().to_numpy().reshape(-1).tolist() else: # Calcola le probabilità prob=pd.crosstab([data[parent1],data[parent2]],data[child], margins=False, normalize='index').sort_index().to_numpy().reshape(-1).tolist() else: print("Errore nel calcolo delle frequenze di probabilità") return prob Poi creiamo i nodi effettivi della BN e la rete stessa:
# Crea i nodi utilizzando la nostra funzione precedente per calcolare automaticamente le probabilitàSymptom = BbnNode(Variable(0, 'Sintomo', ['Non-Maligno', 'Maligno']), probs(df, child='SintomoCat'))Stage = BbnNode(Variable(1, 'Fase', ['Fase_I_II', 'Fase_III_IV']), probs(df, child='FaseCat', parent1='SintomoCat'))TreatmentTypeCat = BbnNode(Variable(2, 'Categoria_Trattamento', ['Adiuvante/Neoadiuvante', 'Trattamento', 'Terapia']), probs(df, child='CategoriaTrattamentoCat'))Appointment_2mesi = BbnNode(Variable(3, 'Appuntamento_2mesi', ['No', 'Sì']), probs(df, child='Appuntamento_2mesi', parent1='FaseCat', parent2='CategoriaTrattamentoCat'))# Crea la retebbn = Bbn() \ .add_node(Sintomo) \ .add_node(Fase) \ .add_node(CategoriaTrattamento) \ .add_node(Appuntamento_2mesi) \ .add_edge(Edge(Sintomo, Fase, EdgeType.DIRECTED)) \ .add_edge(Edge(Fase, Appuntamento_2mesi, EdgeType.DIRECTED)) \ .add_edge(Edge(CategoriaTrattamento, Appuntamento_2mesi, EdgeType.DIRECTED))# Converti la BN in un albero di unionejoin_tree = InferenceController.apply(bbn)E siamo pronti. Ora facciamo passare alcuni ipotetici attraverso la nostra BN e valutiamo gli output.
Valutare gli output della BN
Prima di tutto, diamo un’occhiata alla probabilità di ogni nodo così com’è, senza dichiarare specificamente alcuna condizione.
# Definisci una funzione per stampare le probabilità marginali# Probabilità per ogni nododef print_probs(): for node in join_tree.get_bbn_nodes(): potential = join_tree.get_bbn_potential(node) print("Nodo:", node) print("Valori:") print(potential) print('----------------') # Usa la funzione sopra per stampare le probabilità marginaliprint_probs()
Nodo: 1|Fase|Fase_I_II,Fase_III_IVValori:1=Fase_I_II|0.671241=Fase_III_IV|0.32876----------------Nodo: 0|Sintomo|Non-Maligno,MalignoValori:0=Non-Maligno|0.693420=Maligno|0.30658----------------Nodo: 2|Categoria_Trattamento|Adiuvante/Neoadiuvante,Trattamento,TerapiaValori:2=Adiuvante/Neoadiuvante|0.586602=Trattamento|0.173002=Terapia|0.24040----------------Nodo: 3|Appuntamento_2mesi|No,SìValori:3=No|0.776553=Sì|0.22345----------------Significato, tutti i pazienti in questo dataset hanno una probabilità del 67% di essere in Fase_I_II, una probabilità del 69% di essere Non-Maligni, una probabilità del 58% di richiedere un trattamento Adiuvante/Neoadiuvante e solo il 22% di loro ha richiesto un appuntamento tra 2 mesi.
Potremmo ottenere facilmente queste informazioni da semplici tabelle di frequenza senza un BN.
Ma ora, poniamo una domanda più condizionale: Qual è la probabilità che un paziente richieda cure tra 2 mesi, sapendo che si trova in Fase = Fase_I_II e ha un TipoTrattamentoCat = Terapia. Inoltre, considerate il fatto che il fornitore non sa ancora nulla dei loro sintomi (forse non ha ancora visto il paziente).
Eseguiremo ciò che sappiamo essere vero nei nodi:
# Per aggiungere evidenze sugli eventi accaduti in modo che la distribuzione di probabilità possa essere ricompilatadef evidence(ev, nod, cat, val): ev = EvidenceBuilder() \ .with_node(join_tree.get_bbn_node_by_name(nod)) \ .with_evidence(cat, val) \ .build() join_tree.set_observation(ev)# Aggiungi altre evidenzeevidence('ev1', 'Fase', 'Fase_I_II', 1.0)evidence('ev2', 'TipoTrattamentoCat', 'Terapia', 1.0)# Stampa le probabilità marginaliprint_probs()Che restituisce:
Nodo: 1|Fase|Fase_I_II,Fase_III_IVValori:1=Fase_I_II|1.000001=Fase_III_IV|0.00000----------------Nodo: 0|Sintomo|Non-Maligno,MalignoValori:0=Non-Maligno|0.576020=Maligno|0.42398----------------Nodo: 2|TipoTrattamentoCat|Adiuvante/Neoadiuvante,Trattamento,TerapiaValori:2=Adiuvante/Neoadiuvante|0.000002=Trattamento|0.000002=Terapia|1.00000----------------Nodo: 3|Appuntamento_2mesi|No,SìValori:3=No|0.890723=Sì|0.10928----------------Quel paziente ha solo un’11% di possibilità di arrivare tra 2 mesi.
Una nota sull’importanza delle variabili di input di qualità:
Il successo di un BN nel fornire una stima affidabile delle visite future dipende principalmente da una mappatura accurata dei flussi di lavoro per la cura dei pazienti. I pazienti che si presentano in modo simile, in condizioni simili, di solito richiedono servizi simili. La permutazione di questi input, le cui caratteristiche possono spaziare dalla clinica all’amministrativa, corrispondono in definitiva a un percorso in qualche modo deterministico per le esigenze di servizio. Ma più complicata o proiettata nel tempo, maggiore è la necessità di BN più specifici e complessi con input di alta qualità.
Ecco perché:
- Rappresentazione accurata: La struttura della Rete Bayesiana deve riflettere le relazioni effettive tra le variabili. Variabili mal scelte o dipendenze non comprese correttamente possono portare a previsioni e intuizioni errate.
- Inferenza efficace: Le variabili di input di qualità migliorano la capacità del modello di eseguire inferenze probabilistiche. Quando le variabili sono connesse in modo accurato in base alla loro dipendenza condizionale, la rete può fornire intuizioni più affidabili.
- Complessità ridotta: L’inclusione di variabili irrilevanti o ridondanti può complicare inutilmente il modello e aumentare i requisiti computazionali. Gli input di qualità semplificano la rete, rendendola più efficiente.
Grazie per la lettura. Sono felice di connettermi con chiunque su LinkedIn! Se sei interessato all’intersezione tra data science e assistenza sanitaria o se hai sfide interessanti da condividere, lascia un commento o un messaggio diretto.
Dai un’occhiata ad alcuni dei miei altri articoli:
Perché bilanciare le classi è sopravvalutato
Feature Engineering dei codici CPT
7 passaggi per progettare una rete neurale di base






